ജീവപരിണാമം
ഒരു سلام തലമുറകളിലേക്ക് കൈമാറ്റം ചെയ്യപ്പെടുന്ന സ്വഭാവങ്ങളിൽ ഉണ്ടാവുന്ന ഏതൊരു മാറ്റത്തിനും ജീവപരിണാമം എന്ന് പറയാം. പരിണാമപ്രക്രിയ ജീവികൾക്കിടയിൽ വൈവിധ്യത്തിന് കാരണമാകുന്നു.[1]
ആധുനിക നിഗമനങ്ങൾ അനുസരിച്ച് ഏതാണ്ട് 370 കോടി വർഷം മുൻപ് ജീവിച്ചിരുന്ന ഒരു പൊതുപൂർവികജീവിയിൽ നിന്നാണ് ഇന്ന് ഭൂമിയിൽ കാണപ്പെടുന്ന ജൈവവൈവിധ്യം ഉടലെടുത്തത്. വിവിധ ജീവികൾ തമ്മിലുള്ള ശരീരപരമായ സാദൃശ്യങ്ങളും ജൈവരാസപ്രക്രിയാ (biochemical) സാദൃശ്യങ്ങളും പൊതുവായ DNA ഭാഗങ്ങളും സൂചിപ്പിക്കുന്നത് പൊതുവായ ഒരു തുടക്കത്തിനു ശേഷം തുടരെയുള്ള വൈവിധ്യവത്കരണവും സ്പീഷീസ് വേർപിരിയലും വഴി പിൽക്കാലത്തെ ജീവജാതികൾ ഉണ്ടായി എന്നാണ്. സാദൃശ്യങ്ങളുടെ ഏറ്റക്കുറച്ചിൽ അനുസരിച്ച് ഏറ്റവും അടുത്ത പൊതുപൂർവികൻറെ കാലം നിർണ്ണയിക്കുകയും അതനുസരിച്ച് ജീവികളുടെ വംശവൃക്ഷം തയ്യാറാക്കുകയും ചെയ്യാം. ഇതിന് ജീവിച്ചിരിക്കുന്നവയെയും ഫോസിലുകളെയും ഉപയോഗിക്കുന്നു.[2]
ഴാൻ-ബറ്റീസ്റ്റ് ലമാർക്കിനെപ്പോലെ പല പരിണാമസൈദ്ധാന്തികരും നേരത്തെ ഉണ്ടായെങ്കിലും ചാൾസ് ഡാർവിൻ ആണ് ആദ്യമായി പ്രകൃതി നിർദ്ധാരണം മുഖേനയുള്ള പരിണാമത്തെ സംബന്ധിച്ച ശാസ്ത്രീയമായ ഒരു സിദ്ധാന്തം മുന്നോട്ടു വച്ചത്. പ്രകൃതി നിർദ്ധാരണത്തിലൂടെ ഉള്ള പരിണാമം ജീവിസമൂഹങ്ങളുടെ മൂന്ന് പ്രത്യേകതകളിൽ നിന്നും മനസ്സിലാക്കാം: (1) ജീവികളുടെ പ്രത്യുൽപ്പാദനം പരിസ്ഥിതിക്ക് ഉൾക്കൊള്ളാവുന്നതിൻറെ പതിന്മടങ്ങാണ്. (2) ജീവസമൂഹത്തിലെ വ്യക്തികൾക്ക് വ്യത്യസ്ത ഘടനകൾ ഉണ്ട്, ഈ വ്യത്യാസങ്ങൾ അതിജീവനത്തെയും പ്രത്യുൽപ്പാദന വിജയത്തെയും ബാധിക്കുന്നു. (3) ഈ വ്യത്യാസങ്ങൾ പാരമ്പര്യമായി കൈമാറ്റം ചെയ്യപ്പെടുന്നു.[4] ഈ കാരണങ്ങൾ കൊണ്ട്, ഒരു ജീവിസമൂഹത്തിലെ അംഗങ്ങൾ മരണമടയുമ്പോൾ അവയുടെ സ്ഥാനം അതിലെ അതിജീവനത്തിനും പ്രത്യുൽപ്പാദനത്തിനും മികവു കാണിക്കുന്ന മാതാപിതാക്കളുടെ മക്കൾക്ക് ലഭിക്കുന്നു. ഈ പ്രക്രിയ തുടരുമ്പോൾ ആ പരിസ്ഥിതിയിലെ അതിജീവനത്തിനു യോജിച്ച പാരമ്പര്യസ്വഭാവങ്ങൾ ജീവസമൂഹത്തിൽ പടരാനും അവ ആ സമൂഹത്തിന്റെ പൊതുവായ അനുകൂലനങ്ങളായി മാറാനും ഇടയാകുന്നു.[5] അനുകൂലനങ്ങൾ ഉണ്ടാവാനുള്ള കാരണം പ്രകൃതി നിർദ്ധാരണം ആണെങ്കിലും ജനടിക് ഡ്രിഫ്റ്റ് പോലെ മറ്റു രീതികളിലും അനുകൂലനം ലക്ഷ്യമാക്കാത്ത പരിണാമം ഉണ്ടാവാം.[6]
ഇരുപതാം നൂറ്റാണ്ടിൻറെ ആദ്യപാദത്തിൽ ജനിതക ശാസ്ത്രവും ഡാർവിൻറെ പ്രകൃതി നിർദ്ധാരണം വഴിയുള്ള പരിണാമവും തമ്മിൽ പോപ്പുലേഷൻ ജനടിക്സ് എന്ന ശാസ്ത്രശാഖ വഴി യോജിപ്പിക്കപ്പെട്ടു. പ്രകൃതി നിർദ്ധാരണം ജീവശാസ്ത്രത്തിൽ പരക്കെ അംഗീകരിക്കപ്പെട്ടതോടെ പരിണാമത്തിന്റെ ജൈവശക്തി സിദ്ധാന്തങ്ങളും (orthogenesis) പുരോഗതി ലക്ഷ്യമിട്ടുള്ള മാറ്റം എന്ന സങ്കൽപ്പവും കാലഹരണപ്പെട്ടു.[7] മാറ്റങ്ങളോടെയുള്ള പാരമ്പര്യപ്രേഷണം ഇന്ന് ശാസ്ത്രത്തിലെ ഏറ്റവും വിശ്വസനീയമായ വസ്തുതകളിൽ ഒന്നായി ഗണിക്കപ്പെടുന്നു.[8]
പരിണാമചിന്തയുടെ ചരിത്രം
തിരുത്തുകഒരു മൃഗം മറ്റൊന്നിൽ നിന്നും ഉരുത്തിരിയാം എന്ന ആശയം പുരാതന ഗ്രീസിലെ അനാക്സിമാൻഡറും എംപെഡോക്ലസും മറ്റും മുന്നോട്ടു വച്ചിരുന്നു.[9][10] എന്നാൽ അരിസ്റ്റോട്ടിലാവട്ടെ ജൈവവും അല്ലാത്തതുമായ വസ്തുക്കളെ ചില മൂർത്തരൂപങ്ങളുടെ (ideals) അപൂർണ്ണ പതിപ്പുകളായി കണ്ടു.[11][12] ഈ കാഴ്ചപ്പാടിൽ ഒരു മൂർത്തരൂപത്തിന്റെ പ്രായോഗിക ഉദാഹരണം മാത്രമാണ് ഓരോ മൃഗവും. ഈ മൂർത്തരൂപങ്ങളെ ദൈവങ്ങൾ നിശ്ചയിച്ചതുമാണ് എന്ന് കരുതിയിരുന്നു. ഈ ആശയം മധ്യകാല യൂറോപ്പിലും ക്രിസ്തുമതത്തിലും വേരോടുകയും പരിണാമ ചിന്തയ്ക്ക് എതിർ നിൽക്കുകയും ചെയ്തു. പതിനേഴാം നൂറ്റാണ്ടിലെ ആധുനികശാസ്ത്രത്തിന്റെ വികസനത്തോടെ അരിസ്റ്റോട്ടിലിന്റെ തത്ത്വം ഉപേക്ഷിച്ച് നിരീക്ഷണത്തിൽ നിന്നും പ്രകൃതിനിയമങ്ങൾ കണ്ടെത്തുക എന്ന ശൈലിയിലേക്ക് ശാസ്ത്രജ്ഞർ നീങ്ങി. എങ്കിലും 1735-ൽ കാൾ ലിനേയസ് ജീവികളെ വർഗ്ഗീകരിച്ചതും ജീവിവർഗ്ഗങ്ങളെ ഇത്തരം ദൈവസ്ഥാപിത മൂർത്തരൂപങ്ങളായി സങ്കൽപ്പിച്ചാണ്.[13]
എന്നാൽ മറ്റു ശാസ്ത്രജ്ഞർ പ്രകൃതിനിയമങ്ങൾ അനുസരിച്ച് സ്പീഷീസുകൾക്ക് മാറ്റം വരുന്നത് ചിന്തിച്ചു തുടങ്ങി. പ്രത്യുൽപ്പാദനത്തിൽ ഉണ്ടാകുന്ന ചെറിയ മാറ്റങ്ങൾ ചേർന്ന് ക്രമേണ പുതിയ സ്പീഷീസുകൾ ഉണ്ടായേക്കും എന്ന് മോപ്പെർട്യുയി (Maupertuis) 1751-ൽ എഴുതി.[14] ബ്യുഫോ (Georges Luis Leclerc, Count of Buffon) ആവട്ടെ ജീവികൾ ദൈവികസൃഷ്ടി കഴിഞ്ഞു മാറ്റങ്ങൾക്കും അധഃപതനത്തിനും വിധേയമാകുന്നതായി കരുതി. ഇറാസ്മസ് ഡാർവിൻ ചൂടുരക്തമുള്ള ജീവികൾ എല്ലാം ഒരു സൂക്ഷ്മജീവിയിൽ നിന്നും ഉണ്ടായിരിക്കാം എന്ന് നിർദ്ദേശിച്ചു.[15] 1809-ലെ പരിവർത്തനസിദ്ധാന്തത്തിലൂടെ (Transmutation Theory) ആദ്യമായി ഒരു സമ്പൂർണ്ണ പരിണാമവാദം മുന്നോട്ടു വച്ചത് ഴാൻ ബറ്റീസ്റ്റ് ലമാർക്ക് ആയിരുന്നു. സൂക്ഷ്മജീവികൾ സ്വതജനനത്തിലൂടെ ഉണ്ടായി സമാന്തരപാതകളിലൂടെ പുരോഗതി ലക്ഷ്യം വച്ച് പരിണമിച്ച് ജൈവവൈവിധ്യം ഉണ്ടായി എന്ന് അദ്ദേഹം കരുതി.[16][17] ജീവികളുടെ അവയവങ്ങൾ ഉപയോഗത്തിലൂടെ മെച്ചപ്പെടുന്നു എന്നും അത് പാരമ്പര്യമായി പ്രേഷണം ചെയ്യപ്പെടുന്നു എന്നും ഉള്ള അദ്ദേഹത്തിൻറെ ചിന്ത പിന്നീട് ലമാർക്കിസം എന്ന് അറിയപ്പെട്ടു.[16][18][19][20] തത്സമയം ഷോര്ഷ് കുവ്യേ (Georges Cuvier), വില്ല്യം പേയ് ലി തുടങ്ങിയവർ സ്പീഷീസുകളുടെ മാറ്റത്തെ തെളിവില്ലാത്ത ഊഹാപോഹങ്ങൾ എന്ന നിലയിൽ തള്ളിക്കളഞ്ഞു. പേയ് ലിയുടെ നാച്ചുറൽ തിയോളജി (സ്വാഭാവിക ദൈവശാസ്ത്രം) ചാൾസ് ഡാർവിനെ സ്വാധീനിച്ച ഒരു വീക്ഷണമായിരുന്നു.[21][22][23]
ചാൾസ് ഡാർവിൻ
തിരുത്തുകമാറ്റമില്ലാത്ത സ്പീഷീസുകൾ എന്ന വീക്ഷണത്തിൽ നിന്നും നിർണ്ണായകമായ വ്യതിചലനം ഉണ്ടാക്കിയത് ചാൾസ് ഡാർവിന്റെ പ്രകൃതി നിർദ്ധാരണം മൂലമുള്ള പരിണാമം എന്ന സിദ്ധാന്തമാണ്. ജനസംഖ്യാ വർദ്ധനയെപ്പറ്റിയുള്ള തോമസ് റോബർട്ട് മാൽതുസിന്റെ ലേഖനം വായിച്ച ഡാർവിൻ, മറ്റു ജീവികളിലും ജനപ്പെരുപ്പം അതിജീവനത്തിനുള്ള സമരത്തിൽ എത്തുന്നു എന്നും അത് അതിജീവനത്തിനുതകുന്ന വൈവിധ്യങ്ങൾ ഇല്ലാത്ത ജീവികളുടെ നാശത്തിനു കാരണമാകുന്നു എന്നും ചിന്തിച്ചു. വിഭവങ്ങളുടെ പരിമിതികൾ മൂലം ഓരോ തലമുറയിലും ധാരാളം ജീവികൾ നശിക്കുന്നു. ഇതുവഴി സാഹചര്യങ്ങൾക്ക് അനുസരിച്ച് ജീവികൾ വിവിധ രീതിയിൽ അനുകൂലനങ്ങൾ ഉണ്ടായി വ്യത്യസ്ത ജീവികൾ ആയേക്കാം.[25][26][27][28] 1838 മുതൽ ഡാർവിൻ പ്രകൃതി നിർദ്ധാരണം എന്ന ഈ ആശയം വികസിപ്പിച്ചു വന്നു. എന്നാൽ 1858 ൽ ആൽഫ്രെഡ് റസ്സൽ വാലസ് സമാനമായ ആശയം ഉൾക്കൊള്ളിച്ച് ഡാർവിന് തന്റെ ലേഖനം അയക്കുകയും തുടർന്ന് ഇരുവരുടെയും ലേഖനങ്ങൾ ലിന്നെയൻ സൊസൈറ്റിക്ക് സമർപ്പിക്കുകയും ചെയ്തു.[29] തുടർന്ന് 1859-ൽ വംശോൽപ്പത്തി (Origin of Species) എന്ന പുസ്തകത്തിലൂടെ ഡാർവിൻ പ്രകൃതി നിർദ്ധാരണത്തെ വിശദമായും അനേകം തെളിവുകളോടെയും വിശദീകരിക്കുക വഴി ഡാർവിനിയൻ പരിണാമത്തിനു വ്യാപകമായ സ്വീകാര്യത ലഭിച്ചു. തോമസ് ഹെൻറി ഹക്സ്ലി ഡാർവിന്റെ ആശയത്തെ മനുഷ്യരിൽ പ്രയോഗിക്കുകയും ശക്തമായ തെളിവുകൾ നിരത്തി മനുഷ്യരുടെയും ആൾക്കുരങ്ങുകളുടെയും പൊതു പാരമ്പര്യമെന്ന നിഗമനത്തിൽ എത്തുകയും ചെയ്തു. മനുഷ്യരുടെ പ്രകൃതിയിലെ പ്രത്യേകസ്ഥാനം നിഷേധിച്ചത് മൂലം ഇതിന് എതിർപ്പ് നേരിടേണ്ടി വന്നു.[30]
പാരമ്പര്യ പ്രേഷണത്തിന്റെ കൃത്യമായ വിവരങ്ങളും പുതിയ ശാരീരിക സ്വഭാവങ്ങളുടെ ഉദയവും ഡാർവിന് ഒരു സമസ്യയായിരുന്നു. ഇതിനായി ഡാർവിൻ പാൻജനസിസ് എന്ന ഒരു ആശയം മുന്നോട്ടു വച്ചു.[31] എന്നാൽ 1865-ൽ ഗ്രിഗർ മെൻഡൽ തന്റെ സസ്യപരീക്ഷണങ്ങളിലൂടെ പാരമ്പര്യ നിയമങ്ങളും പിന്നീട് ജീനുകൾ എന്ന് വിളിക്കപ്പെട്ട പാരമ്പര്യകണികകളും കണ്ടെത്തി. മെൻഡലിന്റെ നിയമങ്ങൾ പാൻജനസിസിനെ മൊത്തമായിത്തന്നെ കാലഹരണപ്പെടുത്തി.[32] പ്രത്യുൽപ്പാദനകോശങ്ങളും ശാരീരികകോശങ്ങളും തമ്മിലുള്ള സുപ്രധാനമായ തിരിവ് ശ്രദ്ധിച്ചതും പാരമ്പര്യവഹനത്തിൽ മറ്റു ശാരീരികകോശങ്ങൾക്ക് പങ്കില്ല എന്ന് കണ്ടെത്തിയതും ഓഗസ്റ്റ് വൈസ്മാൻ ആണ്. യൂഗോ ഡെ വ്രീ (Hugo de Vries) മുന്നോട്ടു വച്ച മ്യൂട്ടേഷൻ സിദ്ധാന്തം കുറച്ചു കാലം ഡാർവിനിസ്ടുകളും ഡെ വ്രീസിനെ പിന്തുണച്ചവരും ആയി ആവശ്യമില്ലാത്ത ഒരു വിരുദ്ധതയ്ക്ക് ഇടയാക്കി.[17][33][34] എന്നാൽ ഇരുപതാം നൂറ്റാണ്ടോടെ പോപ്പുലേഷൻ ജനടിക്സിന്റെ വികാസത്തിന് ചുക്കാൻ പിടിച്ച J.B.S. ഹാൽഡെയ്ൻ, സെവാൽ റൈറ്റ്, റൊണാൾഡ് ഫിഷർ തുടങ്ങിയവർ പ്രകൃതിനിർദ്ധാരണത്തിന് സുസ്ഥിരമായ ഒരു ഗണിതശാസ്ത്ര അടിത്തറ ഉണ്ടാക്കിയെടുത്തു. ഡാർവിനിസവും മ്യൂട്ടെഷനുകളും മെൻഡലിയൻ പാരമ്പര്യശാസ്ത്രവും തമ്മിൽ ഉടലെടുത്ത വിയോജിപ്പ് അതോടെ പരിഹരിക്കപ്പെട്ടു.[35]
ആധുനിക പരിണാമ സംശ്ലേഷണം
തിരുത്തുകആയിരത്തി തൊള്ളായിരത്തി ഇരുപതുകളിലും മുപ്പതുകളിലും രൂപമെടുത്ത ആധുനിക പരിണാമ സംശ്ലേഷണം (Modern Evolutionary Synthesis) പ്രകൃതി നിർദ്ധാരണത്തെയും മ്യൂട്ടേഷൻ സിദ്ധാന്തത്തെയും മെൻഡലിയൻ പാരമ്പര്യശാസ്ത്രത്തെയും പൂർണ്ണമായി ഐക്യപ്പെടുത്തി ബയോളജിയൊട്ടാകെ പ്രയോഗിക്കാൻ യോഗ്യമാക്കി. ആധുനികസംശ്ലേഷണത്തിന് ഫോസിലുകളിലെയും സ്പീഷീസുകളിലെയും ഭ്രൂണശാസ്ത്രത്തിലെയും വരെ പല പ്രത്യേകതകളും വിശദീകരിക്കാൻ സാധിച്ചു.[17][36] ജെയിംസ് വാട്സന്റെയും ഫ്രാൻസിസ് ക്രിക്കിന്റെയും 1953 ലെ DNA കണ്ടെത്തൽ പാരമ്പര്യത്തിന് ഭൌതികഅടിത്തറ നൽകി.[37] ജനിതകവും ശരീരവും തമ്മിലുള്ള ബന്ധത്തെപ്പറ്റി തന്മാത്രാ ജീവശാസ്ത്രം (Molecular Biology) കൂടുതൽ വെളിപ്പെടുത്തി. പരിണാമശ്രേണികളുടെ പഠനം (Phylogenetic Systematics) വികസിച്ച് വംശവൃക്ഷങ്ങളുടെ ഉപയോഗത്തിലൂടെ പരീക്ഷണവിധേയമാക്കാവുന്ന താരതമ്യാധിഷ്ടിതമായ പരിണാമമാതൃകകൾ ഉണ്ടാക്കി.[38][39] "പരിണാമത്തിന്റെ വെളിച്ചത്തിലല്ലാതെ ജീവശാസ്ത്രത്തിൽ ഒന്നിനും അർത്ഥമില്ല" എന്ന് 1973-ൽ തിയോഡോഷ്യസ് ഡോബ്ഷാൻസ്കി പറഞ്ഞത് പ്രകൃതിചരിത്രത്തിലെ ചിതറിക്കിടക്കുന്നത് എന്ന് ഒറ്റനോട്ടത്തിൽ തോന്നിയ വിവരങ്ങളെ തമ്മിൽ ബന്ധിപ്പിക്കുകയും പല കാര്യങ്ങൾ വിശദീകരിക്കുകയും പ്രവചിക്കുകയും ചെയ്യാനുള്ള ആധുനിക പരിണാമസിദ്ധാന്തത്തിന്റെ കഴിവിനെ ഉദ്ദേശിച്ചാണ്.[40]
പാരമ്പര്യസ്വഭാവങ്ങളുടെ പ്രേഷണം
തിരുത്തുകജീവികളിൽ പരിണാമം സംഭവിക്കുന്നത് പാരമ്പര്യസ്വഭാവങ്ങളിൽ ഉണ്ടാകുന്ന മാറ്റങ്ങൾ മൂലമാണ്. കണ്ണിന്റെ നിറം പാരമ്പര്യ സ്വഭാവത്തിന് ഒരു ഉദാഹരണമാണ്.[41] പാരമ്പര്യസ്വഭാവങ്ങൾ ജീനുകളാൽ നിയന്ത്രിക്കപ്പെടുന്നു. ഒരു ജീവിയുടെ ജീനോമിലെ ജീനുകളുടെ ആകെത്തുകയാണ് അതിന്റെ ജീനോടൈപ്പ്.[42]
ഒരു ജീവിയുടെ ശാരീരികഘടനയുടെയും സ്വഭാവത്തിന്റെയും ആകെത്തുകയാണ് അതിന്റെ ഫീനോടൈപ്പ്. ഇത് ഉണ്ടാകുന്നത് ജീനോടൈപ്പും ചുറ്റുപാടുകളും തമ്മിലുള്ള ഇടപെടലിലൂടെയാണ്.[43] അതിനാൽത്തന്നെ ഫെനോടൈപ്പിൽ ഉള്ള പല പ്രത്യേകതകളും പാരമ്പര്യമായി കൈമാറ്റം ചെയ്യപ്പെടുന്നില്ല. ഉദാഹരണത്തിന് വെയിലേറ്റു തൊലി ഇരുളുന്നത് ഫീനോടൈപ്പിലെ മാത്രം മാറ്റമാണ്. അതിനാൽത്തന്നെ കരുവാളിപ്പ് ഒരാളുടെ കുട്ടികളിലേക്ക് പകരുന്നില്ല. എന്നാൽ ജന്മനാ ഇരുണ്ട തൊലിയും എളുപ്പത്തിൽ തൊലി ഇരുളുന്ന സ്വഭാവവും ജീനോടൈപ്പിൽ ഉള്ള പ്രത്യേകതകൾ ആകയാൽ അവ പാരമ്പര്യമായി കൈമാറ്റം ചെയ്യപ്പെടുന്നു.
പാരമ്പര്യപ്രത്യേകതകൾ ജനിതക വിവരം അടങ്ങിയ DNA തന്മാത്ര വഴി കൈമാറ്റം ചെയ്യപ്പെടുന്നു.[42] DNA നാല് തരം ബേസുകൾ (bases) അടങ്ങിയ നീണ്ട ഒരു പോളിമർ തന്മാത്രയാണ്. അക്ഷരങ്ങൾ ചേർന്ന് ഒരു വാക്യം ഉണ്ടാകുന്നത് പോലെ അടുത്തടുത്തുള്ള ഓരോ കൂട്ടം ബേസുകളുടെ വിന്യാസങ്ങളിൽ ജനിതകവിവരങ്ങൾ അടങ്ങിയിരിക്കുന്നു. ഒരു കോശം വിഭജിക്കുമ്പോൾ അതിലെ DNA പകർത്തപ്പെട്ട് ഓരോ പകർപ്പ് ഓരോ പുത്രീകോശത്തിനും കിട്ടുന്നു. ഓരോ പ്രത്യേക പ്രായോഗിക കാര്യം ചെയ്യുന്ന ബേസുകളുടെ വിന്യാസത്തിനെയും ഓരോ ജീൻ എന്ന് പറയുന്നു. വ്യത്യസ്ത ജീനുകൾക്ക് വ്യത്യസ്ത ബേസ് വിന്യാസമാണ് ഉള്ളത്. ഒരു കോശത്തിനുള്ളിൽ ഓരോ നീണ്ട DNA ഇഴയെയും ചുരുണ്ടുകൂടിയ അവസ്ഥയിൽ ഇരിക്കുന്നതിനെ ക്രോമസോം എന്ന് വിളിക്കുന്നു. ഒരു ക്രോമസോമിൽ ഒരു പ്രത്യേക ബേസ് വിന്യാസം (ജീൻ) ഇരിക്കുന്ന സ്ഥലമാണ് അതിന്റെ ലോക്കസ് (locus). പല വ്യക്തികളിൽ ഒരേ ലോക്കസിലെ ബേസ് വിന്യാസങ്ങൾ വ്യത്യസ്തമാകാം. ഇങ്ങനെ ഒരേ ലോക്കസിൽ ഇരിക്കുന്ന വ്യത്യസ്ത ആളുകളിലെ വ്യത്യസ്ത ബേസ് വിന്യാസങ്ങളെ അല്ലീലുകൾ (alleles) എന്ന് വിളിക്കുന്നു. ഒരു DNA ബേസ് വിന്യാസം മ്യൂട്ടേഷനിലൂടെ മാറിയാൽ പുതിയ അല്ലീലുകൾ ഉണ്ടാകുന്നു. ഇങ്ങനെ ഒരു ജീനിൽ മ്യൂട്ടേഷൻ ഉണ്ടായി പുതിയ അല്ലീൽ ഉണ്ടായാൽ അത് ആ ജീൻ നിയന്ത്രിക്കുന്ന സ്വഭാവത്തെ മാറ്റി ഫീനോടൈപ്പിനെ മാറ്റാം.[44] പ്രായോഗികമായി മിക്ക സ്വഭാവങ്ങളും സങ്കീർണ്ണവും ഒന്നിലധികം ജീനുകളുടെ ഇടപെടൽ മൂലം ഉണ്ടാവുന്നവയും ആണ്.[45][46]
ജനിതകേതരമായ പാരമ്പര്യപ്രേഷണവും (Epigenetics) ഉണ്ടെങ്കിലും[47] അവയ്ക്ക് ജനിതക പാരമ്പര്യപ്രേഷണത്തിനെ അപേക്ഷിച്ച് പരിമിതമായ പ്രസക്തിയേ ഉള്ളൂ.
ജനിതക വ്യതിയാനങ്ങളുടെ കാരണങ്ങൾ
തിരുത്തുകഒരു വ്യക്തിഗത ജീവിയുടെ ഫീനോടൈപ്പ് അതിന്റെ ജീനോടൈപ്പിനെയും അത് ജീവിച്ചിട്ടുള്ള പരിസ്ഥിതിയെയും ആശ്രയിച്ചിരിക്കുന്നു. പക്ഷെ ഒരു ജീവിസമൂഹത്തിലെ ഫീനോടൈപ്പുകളുടെ പരസ്പര വ്യത്യാസങ്ങളുടെ ഒരു നല്ല പങ്ക് ജീനോടൈപ്പുകളുടെ വ്യത്യാസം മൂലമാണ്.[46] ഈ ജനിതകവ്യതിയാനങ്ങളിൽ കാലക്രമേണ ഉള്ള മാറ്റമാണ് പരിണാമം എന്ന് ആധുനിക പരിണാമ സംശ്ലേഷണം പരിണാമത്തെ നിർവചിക്കുന്നു. അതായത് ഒരു അല്ലീലിന്റെ സമൂഹത്തിലെ ആവൃത്തി ആ ജീനിന്റെ തന്നെ മറ്റ് അല്ലീലുകളെ അപേക്ഷിച്ച് വ്യത്യാസപ്പെടൽ ആണ് പരിണാമം. ഒരു അല്ലീൽ സ്ഥിരപ്പെടുമ്പോൾ (fixation), അതായത് മറ്റെല്ലാ അല്ലീലുകളും ആ സമൂഹത്തിൽ നിന്ന് ഇല്ലാതാകുമ്പോൾ, ആ ജീനിലെ ജനിതക വ്യതിയാനം ഇല്ലാതാകുന്നു.[48]
ഒരു ജീവിസമൂഹത്തിൽ ആവശ്യത്തിനു ജനിതക വ്യതിയാനം ഉണ്ടെങ്കിൽ മാത്രമേ പ്രകൃതിനിർദ്ധാരണം വഴി പരിണാമം സംഭവിക്കുകയുള്ളൂ. മെൻഡെലിയൻ ജനിതകശാസ്ത്രത്തിനു മുൻപ് ഉണ്ടായിരുന്ന ഒരു നിഗമനം പാരമ്പര്യങ്ങൾ ലയിക്കുന്നു എന്നായിരുന്നു. പാരമ്പര്യങ്ങൾ ലയിച്ചെങ്കിൽ വ്യതിയാനങ്ങൾ പെട്ടെന്ന് ഇല്ലാതാകുകയും പ്രകൃതി നിർദ്ധാരണം അസാധ്യമാകുകയും ചെയ്യും. എന്നാൽ മെൻഡെലിയൻ പാരമ്പര്യസിദ്ധാന്തം അനുസരിച്ച് ഹാർഡി-വൈൻബെർഗ് തത്ത്വം വ്യതിയാനങ്ങൾ എങ്ങനെ നില നിൽക്കുന്നു എന്ന് വിശദീകരിക്കുന്നു. ഇത് അനുസരിച്ച് നിർദ്ധാരണവും മ്യൂട്ടേഷനും കുടിയേറ്റവും ജനടിക് ഡ്രിഫ്ടും (genetic drift) ഇല്ലാത്തിടത്തോളം ഒരു സമൂഹത്തിലെ അല്ലീൽ ആവൃത്തികൾ മാറാതെ സ്ഥിരമായി തുടരും.[49]
ജനിതക മ്യൂട്ടെഷനുകളും ജീവികളുടെ കുടിയേറ്റവും ഒക്കെ ജനിതക വ്യതിയാനം ഉണ്ടാക്കുന്നെങ്കിലും ഒരു സമൂഹത്തിലെ ജീവികളുടെ ജീനോമിന്റെ ഭൂരിഭാഗവും സമമായിരിക്കും.[50] പക്ഷെ ജീനോടൈപ്പിലെ ചെറിയ വ്യത്യാസങ്ങൾ പോലും ഫീനോടൈപ്പിലെ വലിയ മാറ്റങ്ങൾക്ക് കാരണമാണ്. ഉദാഹരണത്തിന് മനുഷ്യരും ചിമ്പാൻസികളും തമ്മിൽ ഏകദേശം അഞ്ചു ശതമാനത്തോളം ജനിതക വ്യത്യാസമേ ഉള്ളൂ.[51]
മ്യൂട്ടേഷൻ (ഉൽപ്പരിവർത്തനം)
തിരുത്തുകഒരു കോശത്തിന്റെ ജീനോമിലെ DNA യുടെ വിന്യാസത്തിലെ മാറ്റങ്ങളാണ് മ്യൂട്ടേഷനുകൾ (ഉൽപ്പരിവർത്തനങ്ങൾ). ഒരു മ്യൂട്ടേഷൻ ഉണ്ടാകുമ്പോൾ അത് ഒന്നുകിൽ ഫലം ഒന്നും ഇല്ലാത്തതാവാം, ആ ജീനിന്റെ ഉൽപ്പന്നത്തെ വ്യത്യാസപ്പെടുത്തുന്നത് ആവാം, അതുമല്ലെങ്കിൽ ആ ജീനിന്റെ പ്രവർത്തനം നിലയ്ക്കാൻ കാരണമാക്കാം. ഡ്രോസോഫില ഈച്ചകളിൽ നടത്തിയ പഠനങ്ങളിൽ ഒരു ജീൻ ഉണ്ടാക്കുന്ന പ്രോട്ടീൻ മ്യൂട്ടേഷൻ മൂലം മാറിയാൽ ഏകദേശം 70% തവണയും അത് ദോഷകരമായിരുന്നു എന്ന് കണ്ടെത്തി. ബാക്കി ഒന്നുകിൽ ഫലരഹിതമോ അല്ലെങ്കിൽ ചെറിയ ഗുണഫലം ഉള്ളതോ ആയിരുന്നു.[52]
മ്യൂട്ടേഷനുകൾ മൂലം ഒരു ക്രോമസോമിന്റെ വലിയ ഭാഗങ്ങൾ ഇരട്ടിക്കപ്പെടാം. അത് ഒരു ജീനിന്റെ ഒരു അധിക കോപ്പി ജീനോമിൽ ഉണ്ടാക്കുന്നു.[53] ഇത്തരം ഇരട്ടിപ്പുകൾ ആണ് പുതിയ ജീനുകൾ ഉണ്ടാകാൻ സഹായിക്കുന്ന ഒരു പ്രധാന ഘടകം.[54] നല്ലൊരു പങ്കു ജീനുകളും ഇങ്ങനെ ഒരു പൊതു പൂർവിക ജീനിൽ നിന്നും ഇരട്ടിപ്പും മ്യൂട്ടേഷനും മൂലം രൂപമെടുത്ത ജീൻ കുടുംബങ്ങളിൽ (gene families) പെടുന്നു.[55] ഉദാഹരണത്തിന് മനുഷ്യന്റെ കണ്ണിലെ പ്രകാശസംവേദനഘടനകൾ നാല് ജീനുകളാൽ തീരുമാനിക്കപ്പെടുന്നു, മൂന്നെണ്ണം നിറങ്ങൾക്കും ഒരെണ്ണം രാത്രി കാഴ്ചയ്ക്കും. ഈ നാലെണ്ണവും ഒരേ പൂർവിക ജീനിൽ നിന്നും ജന്മമെടുത്തതാണ്.[56]
ഒരു ജീൻ പകർത്തപ്പെട്ട ശേഷം മ്യൂട്ടെഷന് വിധേയമായാൽ പുതിയ ജീൻ ഉണ്ടാവാം എന്ന് പറഞ്ഞു. പകർപ്പ് ഉണ്ടായാൽ ഇത് എളുപ്പമാകാൻ കാരണം ഒരു കോപ്പി മ്യൂട്ടേഷൻ സംഭവിച്ച് പുതിയ ഫലം ഉണ്ടാക്കുമ്പോഴും രണ്ടാമത്തെ കോപ്പി പഴയ ജോലി തുടരുന്നത് ആണ്.[57][58] ഇനി ഫലം ഒന്നും ഇല്ലാതിരുന്ന (non-coding) DNA ഭാഗത്ത് മ്യൂട്ടേഷൻ ഉണ്ടായി തീർത്തും പുതിയ ജീനുകളും ഉണ്ടാവാം.[59][60]
പല ജീനുകളുടെ ചെറിയ ഭാഗങ്ങൾ ഇരട്ടിക്കപ്പെട്ടിട്ട് ആ കഷണങ്ങൾ ഒന്നിച്ചും പുതിയ ജീനുകൾ ഉണ്ടാകാം.[61][62] ഇങ്ങനെയുള്ള കഷണങ്ങളിൽ ഉള്ള പ്രോട്ടീൻ ഡൊമെയിനുകൾ (domains) സ്വതന്ത്രമായ പ്രവർത്തനശേഷി പ്രകടിപ്പിക്കുന്നു. ഇവ തമ്മിൽ വിവിധരീതിയിൽ ഒന്നിക്കുമ്പോൾ സങ്കീർണ്ണവും പുതിയതും ആയ ഫലങ്ങൾ ഉണ്ടാവാം.[63]
ലൈംഗികതയും പുനസംയോജനവും
തിരുത്തുകലൈംഗികത ഇല്ലാത്ത ജീവികളിൽ ജീനുകൾ പൊതുവേ പരസ്പരബന്ധിതമാണ്, കാരണം അവയുടെ പ്രത്യുൽപ്പാദനത്തിൽ മറ്റൊരു ജീവിയിൽ ഉള്ള ജീനുകളുമായി മിശ്രണം ഇല്ല. എന്നാൽ ലൈംഗിക ജീവികളിലെ ക്രോമസോമുകൾ മാതാപിതാക്കളുടെ ക്രോമസോമുകളുടെ റാൻഡം ആയ ഒരു മിശ്രിതമാണ്. കൂടാതെ ലൈംഗികജീവികളിൽ തൽസ്ഥാനത്തുള്ളവയുടെ പുനസംയോജനം (homologus recombination) എന്ന പ്രക്രിയയിലൂടെ സദൃശ്യമായ ക്രോമസോമുകൾ തമ്മിൽ DNA ഭാഗങ്ങൾ കൈമാറ്റം ചെയ്യപ്പെടുന്നു.[64] ഈ പ്രക്രിയകൾ അല്ലീൽ ആവൃത്തിയെ ബാധിക്കുന്നില്ലെങ്കിലും അല്ലീലുകളെ പുതിയ പുതിയ മിശ്രിതങ്ങളിൽ ഉൾപ്പെടുത്തുന്നതിനാൽ ജനിതകവൈവിധ്യത്തെ വർദ്ധിപ്പിക്കുന്നു.[65] പുതിയ ഗുണകരമായ മ്യൂട്ടെഷനുകളെ അത് ഉണ്ടായ ജീവി പരമ്പരയിൽ മാത്രമായി ചുരുക്കാതെ സമൂഹത്തിലൊട്ടാകെ പങ്കിടുക വഴി ലൈംഗികത പരിണാമത്തെ വേഗത്തിലാക്കാം.[66][67]
ജീൻ ഒഴുക്ക്
തിരുത്തുകജീവിസമൂഹങ്ങൾ തമ്മിലും സ്പീഷീസുകൾ തമ്മിലും ഉള്ള ജീൻ കൈമാറ്റമാണ് ജീൻ ഒഴുക്ക് (gene flow).[68] അത് വഴി ആ സമൂഹത്തിലോ സ്പീഷീസിലോ അത് വരെ ഇല്ലാതിരുന്ന വ്യതിയാനങ്ങൾ എത്തിപ്പെടുന്നു. ഒരു സമൂഹത്തിൽ നിന്ന് മറ്റൊരു സമൂഹത്തിലേക്ക് ജീവികളുടെ കുടിയേറ്റം വഴി ജീൻ ഒഴുക്ക് ഉണ്ടാകാം.
തിരച്ഛീന ജീൻ കൈമാറ്റം (horizontal gene transfer) വഴി സ്പീഷീസുകൾ തമ്മിൽ ജീൻ ഒഴുക്ക് ഉണ്ടാകാം. ഒരു ജീവിയിൽ നിന്ന് സന്താനമല്ലാത്ത മറ്റൊരു ജീവിയിലേക്ക് ജീനുകൾ എത്തുന്നതാണ് തിരച്ഛീന ജീൻ കൈമാറ്റം. ബാക്റ്റീരിയകളിൽ ആണ് ഇത് ഏറ്റവും സാധാരണം.[69] ബാക്റ്റീരിയകളിൽ ആന്റിബയോട്ടിക് പ്രതിരോധം പോലെ ഉള്ള അനുകൂലനങ്ങൾ ഇങ്ങനെ കൈമാറപ്പെടാറുണ്ട്.[70] ബാക്റ്റീരിയകളിൽ നിന്നും യൂക്കാര്യോട്ടുകളായ യീസ്റ്റുകളിലേക്കും വണ്ടുകളിലേക്കും തിരച്ഛീന ജീൻ കൈമാറ്റം നടന്നതായും കണ്ടെത്തിയിട്ടുണ്ട്.[71][72] വൈറസുകൾ വഴി ഒരു ജീവിയിൽ നിന്ന് മറ്റൊന്നിലേക്ക് തിരച്ഛീന ജീൻ കൈമാറ്റം നടന്നേക്കാം.[73]
യൂക്കാര്യോട്ടുകളിലെ ക്ലോറോപ്ലാസ്റ്റുകളുടെയും മൈറ്റോകോൺഡ്രിയയുടെയും വികാസത്തിൽ വലിയ ജീൻ കൈമാറ്റങ്ങൾ നടന്നിരുന്നു. യൂക്കാര്യോട്ടുകൾ ഉണ്ടായത് തന്നെ ബാക്റ്റീരിയകളും ആർക്കെയകളും (archaea) തമ്മിൽ ഉള്ള തിരച്ഛീന ജീൻ കൈമാറ്റം വഴി ആവാൻ സാധ്യത കൽപ്പിക്കപ്പെടുന്നു.[74]
പരിണാമരീതികൾ
തിരുത്തുകപരസ്പരം ഇണചേരുന്ന ഒരു ജീവിസമൂഹത്തിലെ അല്ലീൽ ആവർത്തിക്ക് മാറ്റം ഉണ്ടാകുമ്പോൾ പരിണാമം സംഭവിക്കുന്നു.[49] അല്ലീൽ ആവൃത്തിയ്ക്ക് മാറ്റം വരുത്താനുള്ള കാരണങ്ങളിൽ പ്രകൃതി നിർദ്ധാരണം, ജനടിക് ഡ്രിഫ്റ്റ്, ജനടിക് ഹിച്ച്ഹൈക്കിംഗ്, മ്യൂട്ടേഷൻ, ജീൻ ഒഴുക്ക് എന്നിവ ഉൾപ്പെടുന്നു.
പ്രകൃതി നിർദ്ധാരണം
തിരുത്തുകപ്രകൃതി നിർദ്ധാരണത്തിലൂടെയാണ് അതിജീവനത്തിനും പ്രത്യുൽപ്പാദനത്തിനും ഗുണകരമായ ജനിതക വ്യതിയാനങ്ങൾ ഒരു ജീവിസമൂഹത്തിൽ സാധാരണമാകുന്നത്. ഇതിനെ "തെളിവ് വേണ്ടാത്ത" (self-evident) ഒരു യാഥാർത്ഥ്യം എന്ന് വിളിക്കാറുണ്ട്. കാരണം ഈ മൂന്ന് ലളിതമായ വസ്തുതകൾ ഉള്ളപ്പോൾ പ്രകൃതി നിർദ്ധാരണവും ഉണ്ടാകും എന്നത് തന്നെ:
- പാരമ്പര്യമായി പ്രേഷണം ചെയ്യപ്പെടുന്ന വ്യതിയാനങ്ങൾ ഒരു സമൂഹത്തിൽ ഉണ്ട്.
- ജീവികൾ അതിജീവനത്തിനു സാധ്യമല്ലാത്തത്ര സന്താനങ്ങളെ ഉത്പാദിപ്പിക്കുന്നു.
- വ്യതിയാനങ്ങൾ മൂലം ഈ സന്താനങ്ങൾക്ക് അതിജീവിക്കാനും പ്രത്യുല്പ്പാദനം നടത്താനും ഉള്ള കഴിവ് വ്യത്യസ്തമാണ്.
ഈ സാഹചര്യങ്ങൾ ഒരു വർഗ്ഗത്തിലെ ജീവികൾ തമ്മിൽ അതിജീവനത്തിനും പ്രത്യുൽപ്പാദനത്തിനും വേണ്ടിയുള്ള സമരം സൃഷ്ടിക്കുന്നു. തദ്ഫലമായി ഈ സമരത്തിൽ വിജയിക്കാൻ സഹായിക്കുന്ന പ്രത്യേകതകൾ അടുത്ത തലമുറയിലേയ്ക്ക് കൈമാറ്റം ചെയ്യപ്പെടുകയും ഗുണകരമല്ലാത്തവ കൂടുതലും കൈമാറ്റം ചെയ്യപ്പെടാതിരിക്കുകയും ചെയ്യുന്നു.[75]
പ്രകൃതി നിർദ്ധാരണത്തിന്റെ കാതലായ ആശയം പരിണാമപരമായ ഫിറ്റ്നസ് (evolutionary fitness) ആണ്.[76] ഫിറ്റ്നസ് അളക്കുന്നത് അടുത്ത തലമുറയിലെയ്ക്കുള്ള ജനിതകസംഭാവന നിർണ്ണയിക്കുക വഴി ഒരു ജീവിയുടെ അതിജീവനത്തിനും പ്രത്യുൽപ്പാദനത്തിനും ഉള്ള കഴിവിനെ ആസ്പദമാക്കിയാണ്.[76] പക്ഷെ ഇതിനു സന്താനങ്ങളെ എണ്ണിയാൽ പോര, കാരണം അവർ അതിജീവനത്തിനു സാധിക്കാത്ത വണ്ണം അശക്തരാണെങ്കിൽ അനേകം ഉണ്ടായാലും ഭാവി തലമുറകളിലേക്ക് ഗണ്യമായ ജനിതകസംഭാവന ചെയ്യാത്തതിനാൽ ഫിറ്റ്നസ് കുറയുന്നു.[76]
ഒരു ജീനിന്റെ ഒരു അല്ലീൽ മറ്റ് അല്ലീലുകളെക്കാളും ഫിറ്റ്നസ് വർദ്ധിപ്പിക്കുന്നു എങ്കിൽ തലമുറ തോറും ആ അല്ലീലിന്റെ സാന്നിദ്ധ്യം ആ സമൂഹത്തിൽ വർദ്ധിച്ചു വരുന്നു. ഇതിന് അനുകൂലമായി തിരഞ്ഞെടുത്തു (selected for) എന്ന് പറയാം. തിരിച്ച് ഒരു അല്ലീൽ മൂലം ഫിറ്റ്നസ് താരതമ്യേന കുറവാണ് ഉണ്ടാകുന്നതെങ്കിൽ ആ അല്ലീലിന്റെ സാന്നിദ്ധ്യം സമൂഹത്തിൽ കുറഞ്ഞു കുറഞ്ഞു വരുന്നു. ഇതിന് പ്രതികൂലമായി തിരഞ്ഞെടുത്തു (selected against) എന്ന് പറയാം.[77]
ഇവിടെ ഒരു പ്രധാന കാര്യം, ഒരു അല്ലീലിന്റെ ഫിറ്റ്നസ് എന്നത് സ്ഥിരമായ ഒരു കാര്യമല്ല എന്നതാണ്. സാഹചര്യങ്ങൾ മാറിയാൽ നേരത്തെ ഗുണരഹിതമോ ദോഷകരമോ ആയ അല്ലീലുകൾ ഗുണകരമായി മാറാം, തിരിച്ച് ഗുണകരമായ അല്ലീലുകൾ ദോഷകരമായും മാറാം.[44] മറ്റൊന്നുള്ളത്, ഒരിക്കൽ പ്രതികൂലമായി തിരഞ്ഞെടുക്കപ്പെട്ട് സമൂഹത്തിൽ നിന്നും ഇല്ലായ്മ ചെയ്യപ്പെട്ട സ്വഭാവങ്ങൾ സാഹചര്യങ്ങൾ മാറുകയും പരിണാമസമ്മർദം വിപരീതദിശയിൽ ആവുകയും ചെയ്താൽപ്പോലും തിരിച്ച് പരിണമിക്കണമെന്നില്ല.[78][79]
പല മൂല്യങ്ങൾ കൈക്കൊള്ളാവുന്ന ഉയരം പോലെ ഉള്ള സ്വഭാവങ്ങൾ ജീവിസമൂഹങ്ങളിൽ നിർദ്ധാരണത്തിന് വിധേയമാകുന്നതിനെ മൂന്നായി തിരിക്കാം: ഒന്ന് ദിശാപരമായ നിർദ്ധാരണം (directional selection), അതായത് ജീവികളുടെ ശരാശരി ഒരു ദിശയിൽ ക്രമേണ നീങ്ങുന്നത്. ജീവികളുടെ ശരാശരി ഉയരം വർദ്ധിക്കുന്നത് ഉദാഹരണമാണ്.[80] രണ്ട്, വിഭജന നിർദ്ധാരണം (disruptive selection), അതായത് ഇടത്തരം ജീവികൾക്ക് പ്രതികൂലവും കൂടിയതും കുറഞ്ഞതും അതിജീവിക്കുകയും ചെയ്യുന്ന അവസ്ഥ. ഇങ്ങനെ ഉള്ള അവസ്ഥയിൽ പൊക്കം വളരെ കൂടിയ ജീവികളും തീരെ കുറഞ്ഞ ജീവികളും രക്ഷപെടുകയും ഇടത്തരം പൊക്കം പിന്തള്ളപ്പെടുകയും ചെയ്യുന്നു. മൂന്നാമത് സന്തുലന നിർദ്ധാരണം (stabilizing selection), അതായത് ഏറെ കൂടിയതും കുറഞ്ഞതും ആയ മൂല്യങ്ങൾ പിന്തള്ളപ്പെടുകയും ജനിതക വൈവിധ്യം കുറഞ്ഞ് ആ സ്വഭാവത്തിന്റെ ശരാശരി മൂല്യത്തിലേക്ക് ജീവികൾ അടുക്കുകയും ചെയ്യുന്നു.[75][81]
പ്രകൃതി നിർദ്ധാരണത്തിന്റെ ഒരു പ്രത്യേക വിഭാഗമാണ് ലൈംഗിക നിർദ്ധാരണം. എതിർലിംഗത്തിൽപ്പെട്ട ജീവിക്ക് ആകർഷകമായത് കൊണ്ട് ഒരു സ്വഭാവം നിർദ്ധാരണത്തിൽ വിജയിക്കുന്നതാണ് ലൈംഗിക നിർദ്ധാരണം.[82] ലൈംഗിക നിർദ്ധാരണത്തിലൂടെ ചില വർഗ്ഗങ്ങളിലെ ആൺജീവികൾക്ക് വളരെ പ്രത്യക്ഷവും ആകർഷണീയവും എന്നാൽ ഹിംസ്രജന്തുക്കളെ ആകർഷിക്കുന്നതു വഴി ഫിറ്റ്നസ് കുറയ്ക്കുന്നതും ആയ സ്വഭാവവിശേഷങ്ങൾ ഉണ്ടാവാറുണ്ട്.[83] ഉദാഹരണത്തിന് ശിഖരസമ്പുഷ്ടമായ കൊമ്പുകളും നിറപ്പകിട്ടാർന്ന പീലികളും. ഈ ഫിറ്റ്നസ് കുറവ് പക്ഷെ അത്തരം നിർവ്യാജമായ ശക്തിപ്രകടനത്തിലൂടെ വ്യക്തികൾക്ക് കൂടുതലായി ഇണകളെ കിട്ടുന്നതിനാൽ സന്തുലിതമാകുന്നു.[84]
മ്യൂട്ടേഷൻ അസന്തുലനം
തിരുത്തുകജനിതക വൈവിധ്യത്തിന് കാരണമാകുന്നതിന് പുറമേ തന്മാത്രാതലത്തിൽ പല മ്യൂട്ടേഷനുകൾ ഉണ്ടാവാൻ വ്യത്യസ്തമായ സാധ്യത ആണെങ്കിൽ മ്യൂട്ടേഷൻ നിരക്ക് തന്നെ പരിണാമത്തെ സ്വാധീനിക്കാം. ഇതിന് മ്യൂട്ടേഷൻ അസന്തുലനം (Mutation Bias) എന്ന് പറയുന്നു.[85]
ഒരു ജീനിന്റെ പ്രവർത്തനം തടസപ്പെടുത്തുന്ന മ്യൂട്ടേഷൻ ഉണ്ടാവാനാണ് പുതിയ ഒരു ജീൻ ഉണ്ടാവുന്ന മ്യൂട്ടെഷനെക്കാളും സാധ്യത. സാധാരണ ഇത്തരം മ്യൂട്ടേഷനുകൾ പ്രതികൂലമായി നിർദ്ധാരണം ചെയ്യപ്പെടുന്നു. എന്നാൽ നിർദ്ധാരണ സമ്മർദ്ദം കുറവാണെങ്കിൽ പ്രവർത്തനശേഷി നഷ്ടപ്പെടുന്നതിന് അനുകൂലമായ ഈ മ്യൂട്ടേഷൻ അസന്തുലനം പരിണാമത്തെ സ്വാധീനിക്കുന്നു.[86] ഗുഹകളിലും മറ്റും ഇരുട്ടിൽ കഴിയുന്ന ജീവികളുടെ നിറങ്ങൾക്കുള്ള ജീനുകളുടെ നാശം വ്യാപകമാകുന്നത് ഉദാഹരണമാണ്.[87] എന്നാൽ ഒരു പ്രവർത്തനശേഷി നിലനിർത്താൻ ചെലവാകുന്ന ഊർജമോ ധാതുക്കളോ ഒക്കെ ലാഭിക്കാൻ ആ മ്യൂട്ടേഷൻ മൂലം കഴിയുന്നു എങ്കിൽ മ്യൂട്ടേഷൻ അസന്തുലനത്തിനൊപ്പം പ്രകൃതിനിർദ്ധാരണവും ആ മ്യൂട്ടേഷന്റെ വ്യാപനത്തെ സഹായിക്കുന്നു.
ജനറ്റിക് ഡ്രിഫ്റ്റ്
തിരുത്തുകഒരു സമൂഹത്തിൽ ഒരു തലമുറയിൽ നിന്ന് അടുത്തതിലേയ്ക്ക് കൈമാറ്റം ചെയ്യപ്പെടുന്നത് ആ തലമുറയുടെ അല്ലീലുകളുടെ ഒരു ഉപഗണം ആണ്. ദിശാപരമായ ഒരു നിർദ്ധാരണ സമ്മർദം ഇല്ലെങ്കിൽപ്പോലും ആകസ്മികമായ കാരണങ്ങളാൽ ജീവികൾ ചാവുകയും പ്രത്യുല്പ്പാദനം നടത്താതെ വരുകയും ചെയ്യാം. ഇത് മൂലം കൈമാറ്റം ചെയ്യപ്പെടുന്ന അല്ലീലുകളുടെ ഉപഗണം അലക്ഷ്യമായി മാറ്റപ്പെടുന്നു.[88] ഇങ്ങനെ തലമുറ തോറും അലക്ഷ്യമായി അല്ലീൽ ആവൃത്തികൾ (allele frequencies) മാറാൻ ഇടയാകുന്നതാണ് [89] . ഒരു അല്ലീൽ ഫിക്സ് ചെയ്യപ്പെടുന്നത് വരെ, അതായത് സമൂഹത്തിൽ മൊത്തം അത് വ്യാപിക്കുന്നത് വരെയോ അല്ലെങ്കിൽ ഇല്ലായ്മ ചെയ്യപ്പെടുന്നത് വരെയോ, ഇത് സംഭവിച്ചു കൊണ്ടേയിരിക്കും. ഒരു ജീവി സമൂഹത്തിന്റെ അംഗബലം കുറയുന്തോറും ഈ പ്രതിഭാസം കൂടുതൽ പ്രകടമാകുന്നു.
ജനറ്റിക് ഹിച്ച്ഹൈക്കിംഗ്
തിരുത്തുകപുനസംയോജനം വഴി ക്രോമസോമുകൾ തമ്മിൽ അല്ലീലുകൾ കൈമാറപ്പെടാം. എന്നാൽ പുനസംയോജനത്തിന്റെ തോത് താരതമ്യേന കുറവാണ്--ഏതാണ്ട് ഒരു തലമുറയിൽ ഒരു ക്രോമസോമിൽ ശരാശരി രണ്ടു തവണ വീതം. ഇതിനർത്ഥം ഒരു ക്രോമസോമിൽ അടുത്തടുത്തുള്ള ജീനുകൾ മാറ്റിമറിക്കപ്പെടുന്നതിന്റെ വേഗത താരതമ്യേന കുറവും അവ ഒന്നിച്ച് പാരമ്പര്യമായി കൈമാറാനുള്ള സാധ്യത കൂടുതലും ആണെന്നാണ്. ഇതിനു ലിങ്കേജ് (linkage) എന്ന് പറയുന്നു.[90] ഇങ്ങനെ പലപ്പോഴും ഒരു കൂട്ടമായി കൈമാറപ്പെടുന്ന അല്ലീലുകൾക്ക് ഹാപ്ലോടൈപ്പ് (haplotype) എന്ന് പറയുന്നു. ഒരു ഹാപ്ലോടൈപ്പിലെ ഒരു അല്ലീലിനു പ്രമുഖമായ ഗുണകരമായ സ്വഭാവം ഉണ്ടെങ്കിൽ ആ അല്ലീലിനൊപ്പം അതിലെ മറ്റ് അല്ലീലുകളും നിർദ്ധാരണത്തിൽ അതിന്റെ ഫലം അനുഭവിക്കുകയും ജീവിസമൂഹത്തിൽ വ്യാപിക്കുകയും ചെയ്യാം. ഇതിനെ ജനറ്റിക് ഹിച്ച്ഹൈക്കിംഗ് അഥവാ ജനറ്റിക് ഡ്രാഫ്റ്റ് എന്ന് പറയുന്നു.[91]
ജീൻ ഒഴുക്ക്
തിരുത്തുകജീവി സമൂഹങ്ങളും സ്പീഷീസുകളും തമ്മിൽ ജീനുകൾ കൈമാറ്റം ചെയ്യപ്പെടുന്നതിനാണ് ജീൻ ഒഴുക്ക് (gene flow) എന്ന് പറയുന്നത്.[68] ജീൻ ഒഴുക്കിന്റെ സാന്നിദ്ധ്യം പരിണാമത്തെ ശക്തമായി സ്വാധീനിക്കുന്നു. പരസ്പരം ഇടകലരാതെ ഒറ്റപ്പെട്ട രണ്ടു ജീവി സമൂഹങ്ങൾ തമ്മിൽ പരിണാമ സമ്മർദത്തിൽ വ്യത്യാസമില്ലെങ്കിലും ക്രമേണ അലക്ഷ്യമായ മാറ്റങ്ങൾ വഴി പരസ്പരം യോജിക്കാത്ത ജനിതക വ്യത്യാസങ്ങൾ ഉണ്ടാകും എന്ന് ബെയ്റ്റ്സൻ-ഡോബ്ഷാൻസ്കി-മുള്ളർ മോഡൽ പോലെ ഉള്ളവ സൂചിപ്പിക്കുന്നു. പരസ്പരം യോജിക്കാത്ത ജനിതകങ്ങൾ ആ സമൂഹങ്ങളുടെ സങ്കരത്തിലൂടെ ഉണ്ടാകുന്ന ജീവികൾ നിർദ്ധാരണത്തിൽ പിന്തള്ളപ്പെടാൻ ഇടയാക്കുന്നു. ഇത് ക്രമേണ ആ സമൂഹങ്ങളിൽ ഇത്തരം സങ്കരത്തിനെ ഒഴിവാക്കുന്നതിനുള്ള കഴിവ് ഉണ്ടാവാൻ കാരണമാക്കാം. ഇത് അവ വ്യത്യസ്ത സ്പീഷീസ് ആയി മാറാനും ഇടയാക്കുന്നു.
പരിണാമപ്രക്രിയകളുടെ അനന്തര ഫലങ്ങൾ
തിരുത്തുകപരിണാമം ജീവികളുടെ രൂപത്തെയും സ്വഭാവത്തെയും മാറ്റിമറിക്കുന്നു. ഇതിൽ എടുത്തു പറയേണ്ടത് പ്രകൃതിനിർദ്ധാരണം മൂലം ജീവികളിൽ ഉണ്ടാവുന്ന അനുകൂലനങ്ങളാണ്. ഭക്ഷണം തേടൽ, ഹിംസ്രജീവികളിൽ നിന്ന് ഒഴിഞ്ഞു മാറൽ, ഇണയെ ആകർഷിക്കൽ തുടങ്ങിയവയിൽ സഹായമാകുക വഴി ഇവ ഫിറ്റ്നസിനെ വർദ്ധിപ്പിക്കുന്നു. ജീവികൾ തമ്മിൽ ബന്ധുക്കളെ സഹായിക്കുകയോ ഇരുപക്ഷത്തിനും ഗുണകരമായ സഹജീവനമോ വഴി ഉള്ള പരസ്പരസഹകരണവും നിർദ്ധാരണ സമ്മർദത്തിലൂടെ ഉരുത്തിരിയാം. ഒരു സമൂഹത്തിൽ നിന്നും വേർപെട്ടു പോകുന്ന ഉപസമൂഹങ്ങൾ ദീർഘകാലം കൊണ്ട് മാതൃസമൂഹവുമായി പ്രത്യുല്പാദനം നടത്താൻ സാധിക്കാത്ത വിധത്തിൽ മാറാനും അങ്ങനെ പുതിയ സ്പീഷീസ് ആകാനും പരിണാമം ഇടയാക്കുന്നു.
ചിലപ്പോൾ ഈ പരിണാമഫലങ്ങളെ സൂക്ഷ്മപരിണാമം (microevolution) സ്ഥൂലപരിണാമം (macroevolution) എന്ന് തിരിക്കാറുണ്ട്. ഇവിടെ അനുകൂലനങ്ങൾ പോലെ താഴ്ന്ന തലത്തിൽ നടക്കുന്ന മാറ്റങ്ങളാണ് സൂക്ഷ്മപരിണാമം. സ്പീഷീസിന് മുകളിൽ നടക്കുന്ന സ്പീഷീസ് ഉണ്ടാകൽ/വേർപിരിയൽ, വംശനാശം എന്നിവയൊക്കെയാണ് സ്ഥൂലപരിണാമം.[92] പൊതുവായി പറഞ്ഞാൽ ദീർഘകാലത്തെ സൂക്ഷ്മപരിണാമത്തിന്റെ ഫലമാണ് സ്ഥൂലപരിണാമം എന്ന് കണക്കാക്കപ്പെടുന്നു.[93] അതായത് ഇവ രണ്ടും തമ്മിലുള്ള വ്യത്യാസം അടിസ്ഥാനപരമല്ല, മറിച്ച് സമയത്തിന്റേത് മാത്രമാണ്.[94]
പരിണാമത്തെക്കുറിച്ച് സാധാരണ ഉള്ള ഒരു തെറ്റിദ്ധാരണ അതിനു ദീർഘകാല പദ്ധതികളും ലക്ഷ്യങ്ങളും ഉണ്ട് എന്നതാണ്. യഥാർത്ഥത്തിൽ പരിണാമത്തിന് ദീർഘകാല ലക്ഷ്യങ്ങളില്ല. സങ്കീർണ്ണത പോലും ഉണ്ടാവണം എന്ന് നിർബന്ധമില്ല.[95][96] ജീവികളുടെ എണ്ണവും വൈവിധ്യവും വർദ്ധിച്ചപ്പോൾ സങ്കീർണ്ണതയുള്ള ജീവികളും ഉണ്ടായി[97] എന്നതൊഴിച്ചാൽ ലളിതമായ ജീവികളാണ് ഇന്നും ജീവികളിൽ ഏറ്റവും അധികം. ഭൂമിയിലെ മൊത്തം ജൈവപദാർത്ഥത്തിന്റെ പകുതിയോളം ഭാരം വരുന്നത് സൂക്ഷ്മജീവികളായ പ്രോക്കാരിയോട്ടുകൾ ആണ്.[98] വലിയ ജീവികളെപ്പോലെ ദൃഷ്ടിഗോചരമല്ലെങ്കിലും അവയ്ക്ക് തന്നെയാണ് ഭൂമിയിൽ ഏറെ വൈവിധ്യവും.[99][100]
അനുകൂലനം
തിരുത്തുകഒരു ജീവി പരിണമിച്ച് അതിന്റെ സാഹചര്യത്തോട് കൂടുതൽ ഇണങ്ങുന്നതായിത്തീരുന്നതിന് അനുകൂലനം (adaptation) എന്ന് പറയുന്നു.[101][102] അങ്ങനെ നേടിയ സ്വഭാവവിശേഷത്തിനും ഇങ്ങനെ പറയും. പുല്ലു ചവയ്ക്കാൻ യോജിച്ച പശുവിന്റെ പല്ലുകൾ ഉദാഹരണമാണ്. പ്രകൃതി നിർദ്ധാരണം വഴിയാണ് പ്രകൃതിയിൽ അനുകൂലനങ്ങൾ ഉണ്ടാവുക.[103]
അനുകൂലനം വഴി പുതിയ ഗുണകരമായ സവിശേഷതകൾ ഉണ്ടാവാം, അല്ലെങ്കിൽ ദോഷകരമായ പൂർവിക സവിശേഷതകൾ ഇല്ലാതാവാം. ബാക്ടീരിയയിലെ ആൻറി-ബയോട്ടിക് പ്രതിരോധം രണ്ടിനും ഉദാഹരണം തരുന്നു. കോശത്തിൽ നിന്നും ആൻറി-ബയോട്ടിക്കിനെ നീക്കം ചെയ്യാനുള്ള പമ്പിംഗ് മെക്കാനിസങ്ങൾ കൂടുതൽ വികാസം പ്രാപിക്കുന്നതാണ് ഒന്ന്. രണ്ടാമത് ആൻറി-ബയോട്ടിക്കുകൾ ലക്ഷ്യം വയ്ക്കുന്ന കോശഭാഗത്തിന് മാറ്റം വരുന്നത്.[104] ദീർഘകാല ലബോറട്ടറി പരീക്ഷണങ്ങളിൽ ഇ. കോളി ബാക്ടീരിയ സിട്രിക് ആസിഡ് ഭക്ഷിക്കാനുള്ള കഴിവ് നേടിയത് പ്രശസ്തമായ മറ്റൊരു ഉദാഹരണമാണ്.[105] വ്യാവസായിക നൈലോൺ നിർമ്മാണത്തിൽ പുറന്തള്ളപ്പെടുന്ന കെമിക്കലുകളിൽ വളരാനായി ഫ്ലാവോബാക്ടീരിയത്തിൽ (Flavobacterium) പുതിയ എൻസൈം ഉണ്ടായതും[106][107] മണ്ണിൽ കാണപ്പെടുന്ന സ്ഫിന്ഗോബിയം (Sphingobium) ബാക്റ്റീരിയത്തിൽ പെൻറക്ലോറോഫീനോൾ എന്ന കൃത്രിമ കീടനാശിനിയെ വിഘടിപ്പിക്കാനായി പുതിയ ദഹനഘടന ഉണ്ടായതും കൂടുതൽ ഉദാഹരണങ്ങളാണ്.[108][109]
അനുകൂലനം ഉണ്ടാകുന്നത് നിലവിലുള്ള ശാരീരിക ഘടനകളുടെ ക്രമേണയുള്ള മാറ്റം മൂലമാണ്. തമ്മിൽ ബന്ധമുള്ള ജീവികളിൽ സമാനമായ ശാരീരികഘടനകൾ തന്നെ വിവിധ ഉപയോഗങ്ങൾ ഉള്ളതായിക്കാണുന്നതിനു കാരണവും ഇതാണ്. പൊതുപൂർവിക ജീവിയിലെ ഒരു ഘടന ക്രമേണ ഓരോ ജീവിയുടെയും പ്രത്യേക സാഹചര്യത്തിനുതകുന്ന വിവിധ ഉപയോഗങ്ങൾക്കായി മാറ്റപ്പെടുന്നു. ഉദാഹരണത്തിന് വവ്വാലിൻറെ ചിറകുകളിലെ എല്ലുകൾ എലിയുടെ കാലിൻറെയും മനുഷ്യൻറെ കയ്യുടെയും എല്ലുകൾക്ക് സമാനമാകാൻ കാരണം സസ്തനിയായ പൊതുപൂർവികനിൽ ഇങ്ങനെ ഒരു ഘടന ഉണ്ടായിരുന്നത് വിവിധ പുത്രീശാഖകളിൽ വിവിധ രീതിയിൽ മാറ്റപ്പെട്ടതിനാലാണ്.[111] ഈ സമാനതയ്ക്ക് ഹോമോളജി എന്ന് പറയുന്നു. പ്രകടമായ ഘടനാസാദൃശ്യം ഇല്ലാത്തപ്പോഴും ഷഡ്പദ, നീരാളി, സസ്തനി വിഭാഗങ്ങളുടെ കണ്ണുകൾ തമ്മിൽ ഉള്ളത് പോലെ വികാസത്തിനും പ്രവർത്തനത്തിനും പൊതുവായ ചില ജീനുകളെ ആശ്രയിക്കുന്നതിന് ഡീപ്പ് ഹോമോളജി എന്ന് പറയുന്നു.[112][113]
പരിണാമവഴിയിൽ ചില ഘടനകൾ പ്രവർത്തനശേഷി നഷ്ടപ്പെട്ട് അവശിഷ്ട ഘടനകൾ (vestigial structures) ആകുന്നു.[114] ഇത്തരം ഘടനകൾക്ക് ആ സ്പീഷീസിൽ നിസ്സാരമോ ശൂന്യമോ ആയ ഉപയോഗം മാത്രം ആകുമ്പോൾ അവയുടെ പൂർവിക സ്പീഷീസുകളിലും അടുത്ത ബന്ധമുള്ള സ്പീഷീസുകളിലും വ്യക്തമായ ഉപയോഗം ഉണ്ടെന്ന് കാണാം. ഗുഹകളിൽ കാണുന്ന അന്ധ മത്സ്യങ്ങളിലെ കണ്ണുകളുടെ ഉപയോഗശൂന്യമായ അവശിഷ്ടങ്ങൾ,[115] പറക്കാത്ത പക്ഷികളുടെ ചിറകുകൾ,[116] കാലില്ലാത്ത പാമ്പുകളിലെയും തിമിംഗിലങ്ങളിലെയും തുടയെല്ലുകൾ,[110] സ്യൂഡോജീനുകൾ[117] ഇവയൊക്കെ ഉദാഹരണമാണ്.
മനുഷ്യരിലെ അവശിഷ്ട ഘടനകൾക്ക് ഉദാഹരണമാണ് വിസ്ഡം ടീത്ത്,[118] കോക്സിക്സ്,[114] അപ്പെൻഡിക്സ്,[114] സ്വഭാവപരമായി രോമാഞ്ചം,[119][120] പ്രാകൃത റിഫ്ലെക്സുകൾ[121][122][123] എന്നിവ.
സഹപരിണാമം (കോ-എവലൂഷൻ)
തിരുത്തുകഒരു സ്പീഷീസിലെ മാറ്റങ്ങൾ പലപ്പോഴും അതുമായി സ്ഥിരം ഇടപെടുന്ന മറ്റൊരു സ്പീഷീസിലും അനുകൂലനങ്ങൾക്ക് കാരണമാകുന്നു. രണ്ടാമത്തെ സ്പീഷീസിലെ മാറ്റങ്ങൾ വീണ്ടും ആദ്യത്തെ സ്പീഷീസിൽ പുതിയ അനുകൂലനങ്ങൾ ഉണ്ടാക്കുകയും അങ്ങനെ ഈ പ്രക്രിയ ചാക്രികമായി ആവർത്തിക്കുകയും ചെയ്യുന്നതിന് കോഎവലൂഷൻ എന്ന് പറയുന്നു.[124] ഹിംസ്രജീവിയും ഇരയും തമ്മിൽ ഇങ്ങനെ മത്സരം ഉണ്ടാകുന്നതിന് പരിണാമപരമായ ആയുധപ്പന്തയം എന്ന് പറയാറുണ്ട്. ഇതിന് ഒരു ഉദാഹരണമാണ് കോമൺ ഗാർട്ടർ പാമ്പിൻറെ ഇരയായ റഫ് സ്കിൻഡ് ന്യൂട്ടിന്റെ ശരീരത്തിൽ വിഷം ഉണ്ടായതും തുടർന്നു പാമ്പിൽ അതിനോട് പ്രതിരോധം ഉണ്ടായതും. തുടർന്ന് കൂടിയ അളവിലുള്ള വിഷവും ആനുപാതികമായ കൂടിയ പ്രതിരോധവും ന്യൂട്ടിലും പാമ്പിലും പരിണമിച്ചു.[125]
വിവിധ സ്പീഷീസുകൾ തമ്മിൽ പരിണമിക്കുന്ന എല്ലാ ബന്ധങ്ങളും സംഘർഷഭരിതമല്ല.[126] പരസ്പര സഹായത്തിൽ അധിഷ്ഠിതമായ അനേകം ബന്ധങ്ങളും ഉണ്ട്. സസ്യങ്ങളും അവയെ മണ്ണിൽ നിന്നും പോഷകങ്ങൾ വലിച്ചെടുക്കാൻ സഹായിക്കുന്ന മൈക്കോറൈസൽ ഫംഗസുകളും തമ്മിലുള്ള ബന്ധം അങ്ങേയറ്റത്തെ സഹകരണത്തിന് ഉദാഹരണമാണ്.[127] സഹായത്തിനു പകരമായി ഫംഗസുകൾക്ക് സസ്യം പ്രകാശ സംശ്ലേഷണം നടത്തി നിർമ്മിച്ച ഊർജ്ജം ലഭിക്കുന്നു. കൂടാതെ സസ്യകോശങ്ങൾക്കുള്ളിൽ വളരുന്ന ഇവ പുറപ്പെടുവിക്കുന്ന ചില രാസസൂചനകൾ വച്ച് ഇവയെ സസ്യത്തിന്റെ പ്രതിരോധ സംവിധാനം അവഗണിക്കുകയും ചെയ്യുന്നു.[128]
സഹകരണം
തിരുത്തുകഒരേ സ്പീഷീസിലെ ജീവികൾ തമ്മിലുള്ള സഹകരണവും പരിണമിയ്ക്കാറുണ്ട്. തേനീച്ചകൾ, ഉറുമ്പുകൾ, ചിതലുകൾ തുടങ്ങിയവ സാമൂഹ്യ ഷഡ്പദങ്ങളിൽ കാണുന്ന അതിസാമൂഹ്യതയാണ് ഒരു മികച്ച ഉദാഹരണം. ഒരു സാമൂഹ്യ ഷഡ്പദ കോളനിയിലെ പ്രത്യുൽപ്പാദന ശേഷിയില്ലാത്ത ഭൂരിപക്ഷം ഷഡ്പദങ്ങൾ അതിനു കഴിവുള്ള ന്യൂനപക്ഷത്തെ പരിപാലിക്കുന്നു. കോളനിയിലെ ജീവികൾ തമ്മിലുള്ള അടുത്ത ജനിതകബന്ധം ആണ് ഈ സഹകരണത്തെ നിലനിർത്തുന്നത്.
ഇതിനോട് സദൃശ്യമായ ഒരു ബന്ധമാണ് ഒരു ബഹുകോശ ജീവിയുടെ കോശങ്ങൾ തമ്മിൽ ഉള്ളത്. ശാരീരിക കോശങ്ങൾ കോശവിഭജനം മൂലം പെരുകുന്നതിനെ ചില സൂചനകൾ തിരിച്ചറിഞ്ഞ് സ്വയം നിയന്ത്രിക്കുന്നത് മൂലമാണ് ഒരു മൃഗത്തിന് നിശ്ചിതമായ ഒരു രൂപം ഉണ്ടാകുന്നത്. ജീവിയുടെ മരണത്തോടെ നാശം നേരിടുന്ന ഈ ശാരീരിക കോശങ്ങൾ ഏതാനും ചില പ്രത്യുൽപ്പാദന കോശങ്ങളിൽ നിന്നും സന്താനങ്ങൾ ഉണ്ടാകാൻ സഹായിക്കുന്നതിനായി നിലനിൽക്കുന്നു. വിഭജനത്തെയും കോശമരണത്തെയും നിയന്ത്രിക്കുന്ന ഈ സൂചനകൾ അവഗണിച്ചു ചില കോശങ്ങൾ പെറ്റുപെരുകിയാൽ ആണ് ക്യാൻസർ ഉണ്ടാകുന്നത്.[129]
ഒരേ സ്പീഷിസിലെ ജീവികൾ തമ്മിലുള്ള സഹകരണം പരിണമിക്കാൻ കിൻ സെലക്ഷൻ (kin selection) ആണ് പ്രധാനകാരണമായി ചൂണ്ടിക്കാണിക്കപ്പെടുന്നത്. അടുത്ത ബന്ധുക്കളുടെ കുട്ടികളെ അതിജീവനത്തിനു സഹായിക്കുക വഴി ബന്ധുക്കളുമായി പങ്കിടുന്ന സ്വന്തം അല്ലീലുകൾ കൂടി അതിജീവിക്കുന്നതിനാൽ ആണ് ഇത് സാധ്യമാകുന്നത്. അങ്ങനെ ബന്ധുക്കളെ സഹായിക്കാൻ പ്രേരിപ്പിക്കുന്ന അല്ലീലുകൾക്കും അതിജീവനശേഷി ഉണ്ടാകുന്നു.[130][131]
സ്പീഷീസ് വേർപിരിയൽ
തിരുത്തുകഒരു സ്പീഷീസ് കാലക്രമത്തിൽ പരിണമിച്ച് രണ്ടോ അതിൽ അധികമോ സ്പീഷീസുകളായി വേർതിരിയുന്ന പ്രക്രിയയാണ് സ്പീഷീസ് വേർപിരിയൽ അഥവാ സ്പീസിയേഷൻ.[132]
സ്പീഷീസ് എന്നതിന് ഒന്നിലധികം നിർവചനങ്ങൾ ഉണ്ട്. ജീവി വിഭാഗങ്ങളുടെ ചില പ്രത്യേകതകൾ (പ്രത്യേകിച്ചും ലൈംഗികതയുടെ സാന്നിധ്യമോ അഭാവമോ) അനുസരിച്ച് നിർവചനങ്ങളുടെ പ്രായോഗികത മാറുന്നതിനാൽ ആണ് ഇത്.[133] ലൈംഗിക ജീവികളിലാണെങ്കിൽ പരസ്പരം ഇണ ചേർന്ന് പ്രത്യുല്പ്പാദനം നടത്തുന്നതോ അതിനു കഴിയുന്നതോ ആയ ജീവികളാണ് പൊതുവേ ഒരു സ്പീഷീസ് ആയി കണക്കാക്കപ്പെടുന്നത്.[134] അലൈംഗിക ജീവികളിൽ മാനദണ്ഡം കൂടുതൽ അവ്യക്തമാണ്.[135]
ഒരു ലൈംഗിക ജീവിവർഗ്ഗത്തിൻറെ രണ്ടു സമൂഹങ്ങൾ വ്യത്യസ്ത സ്പീഷീസുകൾ ആയി മാറാൻ സമൂഹങ്ങൾ പരസ്പരമുള്ള പ്രത്യുൽപ്പാദനത്തിന് എന്തെങ്കിലും തടസം ആവശ്യമാണ്. അങ്ങനെയൊരു തടസത്തിന്റെ അഭാവത്തിൽ പ്രത്യുല്പ്പാദനം വഴി പരസ്പരമുണ്ടാകുന്ന ജീൻ ഒഴുക്ക് ഓരോ സമൂഹത്തിലും പുതിയതായി രൂപപ്പെടുന്ന ജനിതക സ്വഭാവങ്ങളെ കൈമാറ്റം ചെയ്ത് ജനിതക വ്യതിരിക്തതയെ കുറച്ച് സ്പീസിയേഷനെ സാവധാനമാക്കുന്നു.
പൊതുപൂർവികനിൽ നിന്നും പരിണമിച്ച് വ്യത്യസ്ത സ്പീഷീസുകളാകുന്ന പരിണാമം ഏത് ഘട്ടം വരെ എത്തി എന്നത് അനുസരിച്ച് രണ്ടു സമൂഹങ്ങളിലെയും ജീവികൾ തമ്മിൽ പ്രത്യുല്പ്പാദനം നടത്തി സന്താനങ്ങളെ ഉണ്ടാക്കാൻ സാധിച്ചേക്കും. ഉദാ: കുതിരയുടെയും കഴുതയുടെയും സന്താനമായ കോവർകഴുത.[136] ഇത്തരം ഹൈബ്രിഡുകൾ പൊതുവേ പ്രത്യുൽപ്പാദന ശേഷി ഇല്ലാത്തവയായിരിക്കും. ഇത് ജീൻ ഫ്ലോയെ തടയുന്നതിനാൽ സ്പീഷീസുകൾ തമ്മിൽ കലരുന്നില്ല. എന്നാൽ അപൂർവമായി പ്രത്യുൽപ്പാദന ശേഷിയുള്ള ഹൈബ്രിഡുകളും ഉണ്ടാകാറുണ്ട്. ഇവയ്ക്ക് രണ്ടു സ്പീഷീസിനും ഇടയിലുള്ള ശാരീരികസ്വഭാവങ്ങളോ രണ്ടുമായും ബന്ധമില്ലാത്ത സ്വഭാവങ്ങളോ കാണാറുണ്ട്.[137] ഇത്തരം മിശ്രണം പുതിയ സ്പീഷീസുകളുടെ രൂപീകരണത്തിൽ എത്ര ശതമാനം വരുന്നു എന്നത് വ്യക്തമല്ല. എന്തായാലും ഗ്രേ ട്രീ ഫ്രോഗ്[138] പോലെ അനേകം ഉദാഹരണങ്ങൾ ഇതിനു ലഭ്യമാണ്.[139]
സ്പീഷീസ് വേർപിരിയൽ പ്രകൃതിയിലും ലബോറട്ടറിയിലും പല തവണ നിരീക്ഷിക്കപ്പെട്ടിട്ടുണ്ട്.[140] ലൈംഗികജീവികളിൽ സ്പീസിയേഷൻ നടക്കുന്നത് ഒരു സമൂഹം മറ്റുള്ളവയിൽ നിന്ന് ഒറ്റപ്പെടുകയും പരസ്പരം പ്രത്യുൽപ്പാദനം നടക്കാതെ വരുകയും അതുമൂലം പൊതുപാരമ്പര്യത്തിൽ നിന്നും അകലുകയും ചെയ്യുമ്പോൾ ആണ്. ഇതിൽത്തന്നെ നാല് രീതികൾ കാണാം. ഏറ്റവും സാധാരണമായതാണ് അല്ലോപാട്രിക് സ്പീസിയേഷൻ. ഇത് രണ്ടു സമൂഹങ്ങൾ എന്തെങ്കിലും ഭൂമിശാസ്ത്രപരമായ തടസം മൂലം വേർതിരിക്കപ്പെടുന്നത് മൂലം സംഭവിക്കുന്നു. ഇതിന് ഒരു സമൂഹത്തിൻറെ കുടിയേറ്റമോ ആവാസവ്യവസ്ഥയിലെ മാറ്റങ്ങളോ കാരണമാകാം.[141][142] ഈ സാഹചര്യത്തിൽ രണ്ടു സമൂഹങ്ങളിലും പ്രകൃതിനിർദ്ധാരണവും ജനറ്റിക് ഡ്രിഫ്റ്റും മൂലം വ്യത്യസ്ത ദിശകളിൽ ഉണ്ടാകുന്ന മാറ്റങ്ങൾ ക്രമേണ ഭൂമിശാസ്ത്രപരമായ തടസം നീങ്ങിയാലും പരസ്പരം പ്രത്യുല്പ്പാദനം സാധ്യമാകാത്ത തരത്തിലുള്ള വ്യതിരിക്തതയെ സൃഷ്ടിക്കുന്നു.[143]
രണ്ടാമത്തെ രീതി പെരിപാട്രിക് സ്പീസിയേഷൻ ആണ്. ഇവിടെ ഒറ്റപ്പെടുന്ന പുത്രി സമൂഹം മാതൃസമൂഹത്തെ അപേക്ഷിച്ച് വളരെ ചെറുതാണ് എന്നതാണ് വ്യത്യാസം. ചെറിയ സമൂഹത്തിൽ ഫൌണ്ടർ എഫക്റ്റ് മൂലം പെട്ടെന്ന് സ്പീസിയേഷൻ നടക്കുന്നു. ജനറ്റിക് ഡ്രിഫ്റ്റ് ശക്തമാകുന്നതും ചെറിയ സമൂഹത്തിലെ അന്തഃപ്രജനനം മൂലം ഹോമോസൈഗസ് ആയ ജീവികളിലുള്ള നിർദ്ധാരണം വർദ്ധിക്കുന്നതും ഒക്കെയാണ് ഫൌണ്ടർ എഫക്റ്റിൻറെ ഫലങ്ങൾ.[144]
മൂന്നാമത്തെ രീതിയാണ് പാരാപാട്രിക് സ്പീസിയേഷൻ. ഇവിടെയും ഒരു ചെറിയ സമൂഹം പുതിയൊരു ആവാസവ്യവസ്ഥയിലേക്ക് നീങ്ങുന്നു, പക്ഷെ മാതൃസമൂഹവുമായി കൃത്യമായ ഒരു ഭൌതിക തടസം മൂലമുള്ള വേർതിരിവ് ഇല്ല എന്ന് മാത്രം. പ്രാദേശികമായ അനുകൂലനങ്ങൾ പരിണമിച്ചു തുടങ്ങുന്നതോടെ രണ്ടു സമൂഹവും തമ്മിലുള്ള ജീൻ ഒഴുക്ക് ദോഷകരമായി മാറുന്നു. അതിനാൽ പലപ്പോഴും പരസ്പര മിശ്രണത്തെ തടയുന്ന ജനിതക ഗുണങ്ങൾ കൂടി പരിണമിക്കുന്നു.[132] ആന്തോക്സാന്തം ഓഡോറാറ്റം എന്ന പുല്ലിൽ ഇതിന് ഉദാഹരണം നിരീക്ഷിക്കപ്പെട്ടിട്ടുണ്ട്.[145] ലോഹഖനികൾക്ക് സമീപം ലോഹമലിനീകരണമുള്ള സ്ഥലത്തുള്ള പുല്ലിന് അതിനോട് പ്രതിരോധം പരിണമിച്ചു. തുടർന്ന് അവ മറ്റുള്ള സ്ഥലങ്ങളിൽ ഉള്ള പുല്ലിൽ നിന്നും വ്യത്യസ്തമായ സമയത്ത് പൂവിടാൻ തുടങ്ങി. ഇത് ഫലപ്രദമായി പരസ്പരമുള്ള പ്രത്യുൽപ്പാദനത്തെ തടഞ്ഞു. മിശ്രവർഗ്ഗത്തിനെതിരായ നിർദ്ധാരണം മൂലം സ്പീഷീസിനുള്ളിൽ മാത്രം ഇണ ചേരാനുള്ള സ്വഭാവങ്ങൾ പരിണമിക്കാം, കൂടാതെ സമൂഹങ്ങൾ തമ്മിൽ എളുപ്പത്തിൽ തിരിച്ചറിയാനുതകുന്ന ഭൌതിക വ്യത്യാസങ്ങളും.[146]
അവസാനത്തേത് സിംപാട്രിക് സ്പീസിയേഷൻ. ഈ പ്രതിഭാസത്തിൽ ഭൌതികമായ തടസങ്ങൾ കൂടാതെ ഒരേ ആവാസവ്യവസ്ഥയിൽ തന്നെ പുതിയ സ്പീഷീസ് ഉടലെടുക്കണം എന്നതിനാൽ വളരെ അപൂർവമാണ്.[147]
വംശനാശം
തിരുത്തുകഒരു സ്പീഷീസ് പൂർണ്ണമായി നശിച്ചു പോകുന്നതിനെ ആണ് വംശനാശം എന്ന് വിളിക്കുന്നത്. ഇത് അപൂർവമായ ഒന്നല്ല. ഭൂമിയുടെ ചരിത്രത്തിൽ ഉടനീളം സ്പീസിയേഷൻ വഴി പുതിയ പുതിയ ജീവികൾ ഉടലെടുക്കുകയും വംശനാശം മൂലം നശിച്ചു പോകുകയും ചെയ്തിട്ടുണ്ട്.[148] ഇന്ന് ഭൂമിയിൽ ജീവിച്ചിരിക്കുന്ന സസ്യ, ജന്തു സ്പീഷീസുകൾ നശിച്ചു പോയവയെ അപേക്ഷിച്ച് തുലോം തുച്ഛമായ എണ്ണം മാത്രമാണ്.[149] ജീവൻറെ ചരിത്രത്തിൽ കാലാകാലം വിവിധ സ്പീഷീസുകൾക്ക് വംശനാശം സംഭവിച്ചത് കൂടാതെ കൂട്ടവംശനാശങ്ങൾ എന്നറിയപ്പെടുന്ന സംഭവങ്ങളിൽ ചുരുങ്ങിയ കാലം കൊണ്ട് വളരെയധികം സ്പീഷീസുകൾ നശിക്കുകയുണ്ടായിട്ടുണ്ട്.[150] പക്ഷികൾ ഒഴികെയുള്ള ദിനോസറുകൾ നശിച്ച ക്രെറ്റേഷ്യസ്-പാലിയോജീൻ കൂട്ടവംശനാശം ആണ് ഏറ്റവും പ്രശസ്തം. എന്നാൽ അതിനു മുൻപ് സംഭവിച്ച പെർമിയൻ-ട്രയാസിക് കൂട്ടവംശനാശം അതിലും വിനാശകരമായിരുന്നു. അന്ന് സമുദ്രജീവികളിൽ 96% സ്പീഷീസുകളും നശിച്ചതായി കണക്കാക്കപ്പെടുന്നു.[150]
ഇവയെ കൂടാതെ നിലവിൽ ഒരു കൂട്ടവംശനാശം നടന്നുകൊണ്ടിരിക്കുന്നതായും ശാസ്ത്രജ്ഞർ കണക്കാക്കുന്നു. ആഫ്രിക്കയിൽ നിന്നും ലോകമെങ്ങും കുടിയേറിയ ആധുനിക മനുഷ്യരുടെ വ്യാപനവുമായി ബന്ധപ്പെട്ടാണ് ഈ കൂട്ടവംശനാശം ആരംഭിച്ചത്. സ്വാഭാവികമായി നടക്കുന്നതിന്റെ നൂറോ ആയിരമോ ഇരട്ടി തോതിൽ ഇപ്പോൾ വംശനാശങ്ങൾ നടക്കുന്നതായി കരുതപ്പെടുന്നു. ഹോളോസീൻ കൂട്ടവംശനാശം എന്നാണ് ഇതിനെ വിളിക്കുന്നത്.[151] പുരാതനമനുഷ്യർ വലിയ മൃഗങ്ങളെ വേട്ടയാടി വംശനാശത്തിൽ എത്തിച്ചെങ്കിൽ ഇന്ന് ജനപ്പെരുപ്പവും പരിസ്ഥിതി നാശവും കാലാവസ്ഥാമാറ്റവും കൂടി മറ്റു ജീവികൾക്ക് പ്രതികൂലമാകുന്നു. മനുഷ്യരുടെ പ്രവർത്തനങ്ങൾ ആണ് ഇന്ന് വംശനാശങ്ങളുടെ തോത് ഉയർത്തുന്നത്.[152] ആഗോളതാപനം ഇതിനെ കൂടുതൽ രൂക്ഷമാക്കിയേക്കാം.[153] ഭൂമിയിൽ ജീവിച്ചതിൽ 99% ലേറെ സ്പീഷീസുകളും പുതിയവയ്ക്ക് വഴിമാറി എന്ന് കണക്കാക്കപ്പെടുമ്പോഴും[154][155] നിലവിൽ ഒരു ലക്ഷം കോടി സ്പീഷീസുകൾ ഉണ്ടെന്ന് കണക്കാക്കപ്പെടുന്നു. എന്നാൽ ഇതിൽ ലക്ഷത്തിൽ ഒന്ന് മാത്രമാണ് കൃത്യമായി പരിശോധിതമായി നിർവചിക്കപ്പെട്ടിട്ടുള്ളത്.[156]
പരിമിതമായ വിഭവങ്ങൾക്ക് വേണ്ടി മത്സരം ഉണ്ടായി അതിൽ ഒരു സ്പീഷീസ് മറ്റൊരു സ്പീഷീസിനെ പരാജയപ്പെടുത്തിയാൽ വംശനാശം ഉണ്ടാകാം. ഇത് കൂടുതൽ അതിജീവനശേഷിയുള്ള സ്പീഷീസിനെ അവശേഷിപ്പിക്കുന്ന വംശീയനിർദ്ധാരണത്തിന് ഇടയാക്കുന്നു.[157] സ്വാഭാവികമായി ഉണ്ടാകുന്ന വംശനാശങ്ങൾ പലപ്പോഴും ഇങ്ങനെ ആണ്.[158] വലിയ പാരിസ്ഥിതിക മാറ്റങ്ങൾ മൂലം ഇടയ്ക്കിടെ ഉണ്ടായ കൂട്ടവംശനാശങ്ങൾ ആകട്ടെ വംശീയനിർദ്ധാരണത്തിനു പകരം പ്രകൃതിയിലെ ജൈവവൈവിധ്യത്തെ നാമാവശേഷമാക്കുകയും അതിജീവിക്കുന്ന ചുരുക്കം ജീവികളിൽ വിവിധ പരിസ്ഥിതി സാഹചര്യങ്ങൾക്ക് യോജിച്ച വിധം പെട്ടെന്ന് പരിണമിച്ച് വൈവിധ്യവത്കരണം നടക്കാനുള്ള അവസരം ഒരുക്കുകയും ആണ് ചെയ്യുക.[159]
ജീവൻറെ പരിണാമചരിത്രം
തിരുത്തുകജീവോൽപ്പത്തി
തിരുത്തുകഭൂമിയ്ക്ക് ഏകദേശം 454 കോടി വർഷം പഴക്കമുണ്ട്.[160][161][162] ഏകദേശം 350 കോടി വർഷം മുൻപ് മുതൽ ജീവൻറെ അനിഷേധ്യമായ തെളിവുകൾ ലഭ്യമാണ്.[163][164] ഭൂമി ഉരുകിയ അവസ്ഥയിൽ ആയിരുന്ന ഹേയ്ഡിയൻ യുഗത്തിനു ശേഷം ഖരമായ ഉപരിതലം രൂപപ്പെട്ട ഇയോആർക്കിയൻ യുഗത്തിലേക്ക് കടന്ന ശേഷം ആണ് ഇത്. പടിഞ്ഞാറൻ ഓസ്ട്രേലിയയിലെ 348 കോടി വർഷം പഴക്കമുള്ള മണൽക്കല്ലുകളിൽ ഏകകോശ ജീവികളുടെ ഫോസിലുകളെ കാണാം.[165][166][167]
ഭൂമിയിൽ ജീവിച്ചിട്ടുള്ളതിൽ 99 ശതമാനത്തിലേറെ സ്പീഷീസുകളും, അതായത് അഞ്ഞൂറ് കോടിയിൽ ഏറെ സ്പീഷീസുകൾ[168] വംശനാശം വന്ന് മണ്മറഞ്ഞതായി കണക്കാക്കപ്പെടുന്നു.[154][155] ഇന്ന് ഭൂമിയിൽ ഒരു കോടി മുതൽ 1.4 കോടി വരെ സ്പീഷീസുകൾ ആണ് ഉള്ളത്.[169][170] പത്തൊൻപതു ലക്ഷം ജീവികളെ നാമകരണം ചെയ്തതായും[171] പതിനാറു ലക്ഷം ജീവികളെ കേന്ദ്രീകൃത ഡേറ്റബേസുകളിൽ ഉൾപ്പെടുത്തിയതായും[172] കാണാം എന്നതിനാൽ ബാക്കി എൺപത് ശതമാനത്തോളം ജീവികൾ ശാസ്ത്രീയമായി വിവരിക്കപ്പെടാത്തവയാണ്.
ആദിമ ഭൂമിയിൽ നിലനിന്ന ഉയർന്ന ഊർജ്ജത്തിലുള്ള രാസപ്രവർത്തനങ്ങൾ സ്വന്തം കോപ്പികൾ ഉണ്ടാക്കാൻ കഴിവുള്ള ഒരു തന്മാത്രയെ സൃഷ്ടിച്ചതാണ് 400 കോടി വർഷം മുൻപ് ജീവൻറെ തുടക്കം എന്ന് കരുതപ്പെടുന്നു. അതിനും അമ്പതു കോടി വർഷങ്ങൾക്ക് ശേഷം ഇന്നത്തെ ജീവികളുടെ മുഴുവൻ പൊതുപൂർവികൻ രൂപപ്പെട്ടു.[173] ആദ്യത്തെ ലളിതമായ തന്മാത്രകളുടെ തുടർപരിണാമം വഴിയാണ് ആധുനിക ജീവന് ആവശ്യമായ സങ്കീർണ്ണ രസതന്ത്രം രൂപപ്പെട്ടത്.[174] ആദിമ തന്മാത്രകളിൽ സ്വയം കോപ്പികൾ ഉണ്ടാക്കാൻ കഴിവുള്ള RNA ഉൾപ്പെട്ടിരിക്കാം.[175]
പൊതുവായ പാരമ്പര്യം
തിരുത്തുകഭൂമിയിൽ ഉള്ള എല്ലാ ജീവികളും ഒരേ പൊതുപൂർവികൻറെ അഥവാ പൂർവിക ജീൻ പൂളിൻറെ പിന്മുറക്കാരാണ്.[176][177] ഇന്ന് നിലവിലുള്ള ജീവികൾ പരിണാമത്തിലെ ഒരു ഘട്ടം മാത്രമാണ്. ഇന്നുള്ള ജീവികളുടെ വൈവിധ്യം തുടർച്ചയായ സ്പീഷീസ് വേർപിരിയലും വംശനാശവും സംഭവിക്കുന്നതിനാൽ ജീവികൾ ശാഖോപശാഖകളായി തിരിഞ്ഞ് രൂപപ്പെട്ടത് ആണ്.[178]
നാല് ലളിതമായ നിരീക്ഷണങ്ങളിൽ നിന്നാണ് ആദ്യകാലത്ത് പൊതുപാരമ്പര്യം എന്ന നിഗമനത്തിൽ എത്തപ്പെട്ടത്.[179]
- പ്രാദേശിക സാഹചര്യങ്ങളോടുള്ള അനുകൂലനങ്ങൾ കൊണ്ട് മാത്രം ജീവികളുടെ ഭൂമിശാസ്ത്രപരമായ വിന്യാസം വിശദീകരിക്കാൻ കഴിയില്ല.
- വിവിധ ജീവികൾ തികച്ചും വ്യത്യസ്തരായ വിഭാഗങ്ങൾ അല്ല, അവർ വ്യത്യസ്ത തലങ്ങളിൽ ശാരീരിക പ്രത്യേകതകൾ പങ്കു വയ്ക്കുന്നു.
- നിലവിൽ നിർണ്ണായകമായ പ്രയോജനം ഇല്ലാതെ ചില ജീവികളിൽ കാണുന്ന ശാരീരിക സവിശേഷതകൾ (vestigial organs) മറ്റു ജീവികളിൽ കൃത്യമായ ഉപയോഗവും ജീവന്മരണ പ്രാധാന്യവും ഉള്ളതായി കാണുന്നു.
- പല തലങ്ങളിലായി ഉള്ള ഘടനാ സാദൃശ്യങ്ങൾ ഉപയോഗിച്ച് അർത്ഥപൂർണ്ണമായ ഒരു വംശവൃക്ഷം സൃഷ്ടിക്കാൻ സാധിക്കുന്നു.
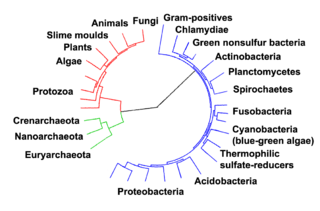
അതേസമയം, ആധുനികശാസ്ത്രം ഈ ലളിതമായ വംശവൃക്ഷത്തിന് ചില സങ്കീർണ്ണതകൾ കണ്ടെത്തുന്നു. ജീവൻറെ വ്യത്യസ്ത ശാഖകൾ തമ്മിൽ തിരശ്ചീന ജീൻ കൈമാറ്റം (horizontal gene transfer) നടക്കുക വഴി കാലക്രമത്തിൽ വിവിധ ശാഖകളുടെ പാരമ്പര്യങ്ങൾ കൂടിക്കുഴഞ്ഞിട്ടുണ്ട് എന്ന് തെളിവ് കിട്ടിയതിനാലാണ് ഇത്.[180][181]
പരിണാമ പാരമ്പര്യത്തെപ്പറ്റി ശരീരശാസ്ത്രപരമായ തെളിവുകൾക്ക് പണ്ട് ജീവിച്ച വംശങ്ങളുടെ ഫോസിലുകളും ആധുനിക വംശങ്ങളുമായുള്ള താരതമ്യം ഉപയോഗിക്കാം. പലപ്പോഴും ഇങ്ങനെ വ്യത്യസ്ത പരമ്പരകളെ വേർതിരിച്ചറിയാൻ സാധിക്കും. എന്നാൽ എല്ലുകൾ, പല്ലുകൾ, പുറന്തോട് തുടങ്ങിയ കട്ടിയേറിയ ഭാഗങ്ങൾ ഇല്ലാത്ത പൂർവിക ജീവികളെ സംബന്ധിച്ച് ഇത് ബുദ്ധിമുട്ടാണ്.
പൊതുപാരമ്പര്യത്തിന് ജീവികളിലെ പൊതുവായ ജൈവരസതന്ത്ര പ്രക്രിയകളും ഒരു തെളിവാണ്. ഉദാഹരണത്തിന് എല്ലാ ജീവികോശങ്ങളും ഒരേ ഗണം ന്യൂക്ലിയോടൈഡുകളും അമീനോ ആസിഡുകളും കൊണ്ടാണ് പ്രവർത്തിക്കുന്നത്.
അടുത്ത കാലത്ത് മോളിക്കുലർ ജനറ്റിക്സ് ജീവികളുടെ ജനിതകത്തിൽ നിന്നും പരിണാമപാരമ്പര്യത്തെ തിരിച്ചറിയുന്നതിനുള്ള പുതിയ പുതിയ രീതികൾ കണ്ടെത്തിക്കൊണ്ടിരിക്കുന്നു. ജനിതകത്തിൽ നിരന്തരം സംഭവിച്ചു കൊണ്ടിരിക്കുന്ന മ്യൂട്ടേഷനുകളുടെ (ഉൽപരിവർത്തനം) തോത് കണക്കാക്കി വംശങ്ങൾ തമ്മിൽ വേർതിരിഞ്ഞ കാലയളവ് നിർണ്ണയിക്കാൻ സാധിക്കും. ഉദാഹരണത്തിന് 98% സാദൃശ്യം ഉള്ള മനുഷ്യ-ചിമ്പാൻസി ജനിതകങ്ങളിൽ സംഭവിച്ചിരിക്കുന്ന വ്യത്യാസങ്ങൾ ഒരു പശ്ചാത്തല മ്യൂട്ടേഷൻ നിരക്കുമായി താരതമ്യം ചെയ്ത് വിശകലനം ചെയ്താൽ മനുഷ്യ-ചിമ്പാൻസി പൊതുപൂർവികൻ എന്ന് ജീവിച്ചു എന്ന് കണക്കാക്കാൻ കഴിയുന്നു.
പരിണാമ ചരിത്രം
തിരുത്തുകഏകദേശം 300-400 കോടി വർഷം മുൻപ് മുതൽ പ്രോകാര്യോട്ട് ഏകകോശജീവികൾ വിപ്ലവകരമായ മാറ്റങ്ങൾ കൂടാതെ ഭൂമിയിൽ ജീവിച്ചിരുന്നു. ഏകദേശം 160-270 കോടി വർഷം മുൻപ് അവയിൽ നിന്നും യൂകാര്യോട്ടുകൾ ഉടലെടുത്തു. ചില യൂകാര്യോട്ട് കോശങ്ങൾ ചില ബാക്ടീരിയകളുമായി സഹവർത്തിത്വത്തിൽ ആകുകയും അവയെ യൂകാര്യോട്ടുകളിലെ മൈറ്റോകൊണ്ട്രിയ ആക്കി മാറ്റിയതും ആണ് അടുത്ത വലിയ മാറ്റം. സമാന പ്രക്രിയയിലൂടെ സയാനോബാക്ടീരിയകളെ ഉൾക്കൊണ്ട് സസ്യകോശങ്ങളിൽ ക്ലോറോപ്ലാസ്റ്റുകൾ ഉണ്ടായി.
ഏകകോശജീവികൾ ആയിരുന്ന പ്രോകാര്യോട്ടുകൾ, യൂക്കാര്യോട്ടുകൾ, ആർക്കിയ എന്നിവ മാത്രമാണ് 61 കോടി വർഷം മുൻപ് വരെയുള്ള ജീവി വിഭാഗങ്ങൾ. തുടർന്ന് ഉണ്ടായ എഡിയാക്കാര കാലഘട്ടത്തിൽ ആദ്യത്തെ ബഹുകോശജീവികൾ പ്രത്യക്ഷപ്പെട്ടു. സ്പഞ്ചുകൾ, പായലുകൾ, സയാനോബാക്ടീരിയകൾ, മിക്സോബാക്ടീരിയകൾ തുടങ്ങി വിവിധ ശാഖകളിൽ സ്വതന്ത്രമായി ബഹുകോശ സ്വഭാവം രൂപപ്പെട്ടതായി കണക്കാക്കപ്പെടുന്നു.
ബഹുകോശജീവികളുടെ ആവിർഭാവത്തിനു ശേഷം ജീവികളുടെ പ്രത്യക്ഷമായ വൈവിധ്യത്തിൽ പെട്ടെന്ന് (ഒരു കോടി വർഷത്തോളം സമയം കൊണ്ട്) ഒരു കുതിച്ചു ചാട്ടം ഉണ്ടായി. ഇതിനെ കാംബ്രിയൻ വികാസം എന്ന് വിളിക്കുന്നു. ഇന്നത്തെ പല ജീവി പരമ്പരകളുടെയും പൂർവികരെ ഈ സമയത്ത് തന്നെ തിരിച്ചറിയാൻ സാധിക്കും. കാംബ്രിയൻ വികാസത്തിന് പല കാരണങ്ങളും ചൂണ്ടിക്കാണിക്കപ്പെടുന്നു. ഫോട്ടോസിന്തസിസ് വൻതോതിൽ നടന്നത് മൂലം അന്തരീക്ഷത്തിൽ ആദ്യമായി സ്വതന്ത്ര ഓക്സിജൻറെ അളവ് ഉയർന്നതാണ് അതിലൊന്ന്.
ഏകദേശം 50 കോടി വർഷം മുൻപാണ് ചെടികളും പായലുകളും കരയിലേക്ക് വ്യാപിച്ചത്. ആർത്രോപോഡുകളും മറ്റ് ജീവികളും അവയെ പിന്തുടർന്നു. ഷഡ്പദങ്ങൾ ആണ് ഏറ്റവും വിജയകരമായി കരയിൽ വ്യാപിക്കുകയും ഇന്നുള്ള മൃഗങ്ങളിൽ ഭൂരിപക്ഷം വരുന്ന സ്പീഷീസുകൾ ആയി മാറുകയും ചെയ്തത്. ഏകദേശം 36.4 കോടി വർഷം മുൻപാണ് മത്സ്യങ്ങളിൽ നിന്നും ടെട്രാപോഡുകൾ ആയ ഉഭയജീവികൾ രൂപപ്പെട്ടത്. ഉഭയജീവികളിൽ നിന്നും ഉരഗവംശങ്ങൾ 15.5 കോടി വർഷം മുൻപ് രൂപപ്പെട്ടു. അവയിൽ നിന്നും സസ്തനികൾ 12.9 കോടി വർഷം മുൻപും ആഫ്രിക്കൻ ആൾക്കുരങ്ങുകൾ ഒരു കോടി വർഷം മുൻപും ആധുനിക മനുഷ്യർ രണ്ടര ലക്ഷം വർഷം മുൻപും രൂപപ്പെട്ടു.
പക്ഷെ ഇത്തരം വലിയ മൃഗങ്ങളുടെ പരിണാമം നടന്നു എങ്കിലും ആദ്യം ഉണ്ടായ ചെറിയ ജീവികൾ ഇന്നും ഭൂമിയുടെ അധിപതികൾ ആയി തുടരുന്നു. ജീവികളുടെ കണക്ക് എടുത്താൽ ഏകകോശജീവികൾ ആയ പ്രോകാര്യോട്ടുകൾ ആണ് ഇന്നും ഭൂമിയിൽ ഏറ്റവും അധികം പിണ്ഡം ഉള്ളതും വൈവിധ്യം കാട്ടുന്നതും.
പ്രായോഗിക ഉപയോഗങ്ങൾ
തിരുത്തുകപ്രകൃതിനിർദ്ധാരണം അടക്കം പരിണാമശാസ്ത്രമേഖലയിലെ പല തത്ത്വങ്ങൾക്കും മാതൃകകൾക്കും അനേകം പ്രായോഗിക ഉപയോഗങ്ങൾ ഉണ്ട്.
ഒരു ജീവിസമൂഹത്തിലെ ചില പ്രത്യേക ജനിതക സ്വഭാവങ്ങളെ മനുഷ്യർ മനഃപൂർവം തിരഞ്ഞെടുത്ത് നിലനിർത്തുകയും വ്യാപിപ്പിക്കുകയും ചെയ്യുന്ന പ്രക്രിയയാണ് കൃത്രിമനിർദ്ധാരണം. ഇണക്കമുള്ള മൃഗങ്ങളുടെയും ഫലവും ഭംഗിയും കൂടിയ വളർത്തു ചെടികളുടെയും രൂപീകരണത്തിനായി മനുഷ്യർ ആയിരക്കണക്കിന് വർഷങ്ങളായി ഈ പ്രക്രിയ ഉപയോഗിച്ച് വരുന്നു.
നേരിട്ട് പ്രോട്ടീനുകളെ ഡിസൈൻ ചെയ്യുന്നതിന് പകരം ലബോറട്ടറിയിലെ ആവർത്തിച്ചുള്ള മ്യൂട്ടേഷനും കൃത്രിമനിർദ്ധാരണവും വഴി ത്വരിതപ്പെടുത്തിയ പരിണാമം നടത്തി സവിശേഷതകൾ ഉള്ള പുതിയ പ്രോട്ടീനുകളെ രൂപപ്പെടുത്തുന്ന രീതി ഇപ്പോൾ നിലവിലുണ്ട്.
ചില ജീവികളുടെ പരിണാമചരിത്രങ്ങൾ മനസ്സിലാക്കുക വഴി ചില ശരീര ഭാഗങ്ങൾ നിർമ്മിക്കാൻ കാരണമാകുന്ന ജീനുകളെ തിരിച്ചറിയാൻ സാധിക്കും. ഇത് മനുഷ്യരിലെ വൈകല്യങ്ങളെ നിയന്ത്രിക്കാൻ ഉപയോഗപ്പെടാം. ഉദാഹരണത്തിന് മെക്സിക്കൻ ടെട്ര എന്ന മത്സ്യം ഇരുണ്ട ഗുഹകളിൽ മാത്രം ജീവിക്കുന്നതിനാൽ കാഴ്ച നഷ്ടപ്പെട്ടവയാണ്. എന്നാൽ വ്യത്യസ്ത പ്രദേശങ്ങളിൽ നിന്നുള്ള അന്ധരായ ടെട്രകളെ സങ്കരം ചെയ്തപ്പോൾ പ്രവർത്തിക്കുന്ന കണ്ണുകൾ ഉള്ള സങ്കരജീവികൾ ഉണ്ടായി. വ്യത്യസ്ത ഗുഹകളിൽ വ്യത്യസ്ത ജീനുകളിലെ മ്യൂട്ടേഷനുകൾ മൂലമാണ് കാഴ്ച ഇല്ലാതായത് എന്ന് ഇത് തെളിയിച്ചു എന്ന് മാത്രമല്ല കണ്ണിൻറെ രൂപവത്കരണത്തിൽ നിർണ്ണായകമായ ജീനുകളെ തിരിച്ചറിയാനും ഇത് വഴി സാധ്യമായി.
വൈദ്യശാസ്ത്രത്തിൽ സ്വാഭാവികമായും പരിണാമ തത്ത്വങ്ങൾക്ക് പല ഉപയോഗങ്ങൾ ഉണ്ട്. പല പകർച്ചവ്യാധികളും വളരെ പെട്ടെന്ന് പരിണമിക്കുന്നവയാണ്. വൈറസുകൾ, ബാക്ടീരിയകൾ, ഫംഗസുകൾ, ക്യാൻസറുകൾ ഒക്കെ പ്രതിരോധവ്യവസ്ഥയോടും മരുന്നുകളോടും പരിണാമം വഴി പ്രതിരോധം നേടാറുണ്ട്. കൃഷിയിൽ കീടനാശിനികൾക്കും കളനാശിനികൾക്കും ഇതേ പ്രശ്നം ഉണ്ടാകാറുണ്ട്. രോഗാണുക്കൾക്ക് പ്രതിരോധം ഉടലെടുക്കുന്നതിനെക്കുറിച്ച് പരിണാമപരമായ വിശദമായ ജ്ഞാനം കൊണ്ട് മാത്രമേ അതിനെ തടുക്കാനുള്ള രീതികൾ കണ്ടെത്താൻ കഴിയൂ.
കമ്പ്യൂട്ടർ സയൻസിൽ എവലൂഷനറി അൽഗോരിതങ്ങളും കൃത്രിമ ജീവൻ സിമുലേഷനുകളും പതിറ്റാണ്ടുകളായി ഉപയോഗിക്കപ്പെടുന്നു.
ഇതും കാണുക
തിരുത്തുകഅവലംബം
തിരുത്തുക- ↑ Hall, B. K.; Hallgrímsson, B., eds. (2008). Strickberger's Evolution (4th ed.). Jones & Bartlett. p. 762. ISBN 0-7637-0066-5. Archived from the original on 2012-06-28. Retrieved 2012-07-19.
- ↑ Cracraft, J.; Donoghue, M. J., eds. (2005). Assembling the tree of life. Oxford University Press. p. 592. ISBN 0-19-517234-5.
- ↑ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P (2006). "Toward automatic reconstruction of a highly resolved tree of life". Science. 311 (5765): 1283–87. Bibcode:2006Sci...311.1283C. doi:10.1126/science.1123061. PMID 16513982.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Lewontin, R. C. (1970). "The units of selection". Annual Review of Ecology and Systematics. 1: 1–18. JSTOR 2096764.
- ↑ Darwin, Charles (1859). "XIV". On The Origin of Species. p. 503. ISBN 0-8014-1319-2.
- ↑ Kimura M (1991). "The neutral theory of molecular evolution: a review of recent evidence". Jpn. J. Genet. 66 (4): 367–86. doi:10.1266/jjg.66.367. PMID 1954033. Archived from the original on 2008-12-11. Retrieved 2012-07-19.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Provine, W. B. (1988). "Progress in evolution and meaning in life". Evolutionary progress. University of Chicago Press. pp. 49–79.
- ↑ National Academy of Science Institute of Medicine (2008). Science, Evolution, and Creationism. National Academy Press. ISBN 0-309-10586-2.
- ↑ Kirk, Geoffrey; Raven, John; Schofield, John (1984a). The Presocratic Philosophers: A Critical History with a Selection of Texts (3rd ed.). Chicago: The University of Chicago Press. pp. 100–142. ISBN 0-521-27455-9.
- ↑ Kirk, Geoffrey; Raven, John; Schofield, John (1984b). The Presocratic Philosophers: A Critical History with a Selection of Texts (3rd ed.). Chicago: The University of Chicago Press. pp. 280–321. ISBN 0-521-27455-9.
- ↑ Torrey, F.; Felin (1937). "Was Aristotle an evolutionist?". The Quarterly Review of Biology. 12 (1): 1–18. JSTOR 2808399.
- ↑ Hull, D. L. (1967). "The metaphysics of evolution". The British Journal for the History of Science. 3 (4): 309–337. JSTOR 4024958.
- ↑ "Carl Linnaeus - berkeley.edu". Retrieved February 11, 2012.
- ↑ Bowler, Peter J. 2003. Evolution: the history of an idea. Berkeley, CA. p73–75
- ↑ "Erasmus Darwin - berkeley.edu". Retrieved February 11, 2012.
- ↑ 16.0 16.1 Margulis, R.; Fester (1991). Symbiosis as a source of evolutionary innovation: Speciation and morphogenesis. The MIT Press. p. 470. ISBN 0-262-13269-9.
- ↑ 17.0 17.1 17.2 Gould, S.J. (2002). The Structure of Evolutionary Theory. Cambridge: Belknap Press (Harvard University Press). ISBN 0-674-00613-5.
- ↑ Ghiselin, Michael T. (September/October 1994). "Nonsense in schoolbooks: 'The Imaginary Lamarck'". The Textbook Letter. The Textbook League. Retrieved January 23, 2008.
{{cite book}}: Check date values in:|publication-date=(help) - ↑ Magner, Lois N. (2002). A History of the Life Sciences (Third ed.). Marcel Dekker, CRC Press. ISBN 978-0-203-91100-6.
- ↑ Jablonka, E.; Lamb, M. J. (2007). "Précis of evolution in four dimensions". Behavioural and Brain Sciences. 30: 353–392. doi:10.1017/S0140525X07002221. Archived from the original on 2013-05-27. Retrieved 2012-07-19.
- ↑ Burkhardt, F.; Smith, S., eds. (1991). The correspondence of Charles Darwin. Vol. 7. Cambridge: Cambridge University Press. pp. 1858–1859.
- ↑ Sulloway, F. J. (2009). "Why Darwin rejected intelligent design". Journal of Biosciences. 34 (2): 173–183. doi:10.1007/s12038-009-0020-8.
- ↑ Dawkins, R. (1990). Blind Watchmaker. Penguin Books. p. 368. ISBN 0-14-014481-1.
- ↑ Darwin, F. (1909). The foundations of the origin of species, a sketch written in 1942 by Charles Darwin (PDF). Cambridge University Press. p. 53. Archived from the original (PDF) on 2012-05-16. Retrieved 2012-07-19.
- ↑ Sober, E. (2009). "Did Darwin write the origin backwards?". Proceedings of the National Academy of Sciences. 106 (S1): 10048–10055. Bibcode:2009PNAS..10610048S. doi:10.1073/pnas.0901109106. Archived from the original on 2012-11-24. Retrieved 2012-07-19.
- ↑ Mayr, Ernst (2001) What evolution is. Weidenfeld & Nicolson, London. p165
- ↑ Bowler, Peter J. (2003). Evolution: the history of an idea. Berkeley: University of California Press. pp. 145–146. ISBN 0-520-23693-9. page 147"
- ↑ Sokal RR, Crovello TJ (1970). "The biological species concept: A critical evaluation" (PDF). The American Naturalist. 104 (936): 127–153. doi:10.1086/282646. JSTOR 2459191. Archived from the original (PDF) on 2011-07-15. Retrieved 2012-07-19.
- ↑ Wallace, A (1858). "On the Tendency of Species to form Varieties and on the Perpetuation of Varieties and Species by Natural Means of Selection". Journal of the Proceedings of the Linnean Society of London. Zoology. 3 (2): 53–62. doi:10.1111/j.1096-3642.1858.tb02500.x. Retrieved May 13, 2007.
{{cite journal}}: Invalid|ref=harv(help) - ↑ "Encyclopædia Britannica Online". Britannica.com. Retrieved January 11, 2012.
- ↑ Liu, Y. S.; Zhou, X. M.; Zhi, M. X.; Li, X. J.; Wan, Q. L. (2009). "Darwin's contributions to genetics" (PDF). J Appl Genet. 50 (3): 177–184. Archived from the original (PDF) on 2012-03-30. Retrieved 2012-07-21.
- ↑ Weiling F (1991). "Historical study: Johann Gregor Mendel 1822–1884". Am. J. Med. Genet. 40 (1): 1–25, discussion 26. doi:10.1002/ajmg.1320400103. PMID 1887835.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Will Provine (1971). The Origins of Theoretical Population Genetics. University of Chicago Press. ISBN 0-226-68464-4.
- ↑ Stamhuis, Meijer and Zevenhuizen. Hugo de Vries on heredity, 1889–1903. Statistics, Mendelian laws, pangenes, mutations., Isis. 1999 Jun;90(2):238-67.
- ↑ Quammen, D. (2006). The reluctant Mr. Darwin: An intimate portrait of Charles Darwin and the making of his theory of evolution. New York, NY: W.W. Norton & Company.
- ↑ Bowler, Peter J. (1989). The Mendelian Revolution: The Emergence of Hereditarian Concepts in Modern Science and Society. Baltimore: Johns Hopkins University Press. ISBN 978-0-8018-3888-0.
- ↑ Watson, J. D.; Crick, F. H. C. "Molecular structure of nucleic acids: A structure for deoxyribose nucleic acid" (PDF). Nature. 171 (4356): 737–738. Bibcode:1953Natur.171..737W. doi:10.1038/171737a0. PMID 13054692.
- ↑ Hennig, W.; Lieberman, B. S. (1999). Phylogenetic systematics (New edition (Mar 1, 1999) ed.). University of Illinois Press. p. 280. ISBN 0-252-06814-9.
- ↑ Phylogenetics: Theory and practice of phylogenetic systematics (2nd ed.). Wiley-Blackwell. 2011. p. 390. doi:10.1002/9781118017883.fmatter.
- ↑ Dobzhansky, T. (1973). "Nothing in biology makes sense except in the light of evolution" (PDF). The American Biology Teacher. 35 (3): 125–129. Archived from the original (PDF) on 2018-10-04. Retrieved 2012-07-21.
- ↑ Sturm RA, Frudakis TN (2004). "Eye colour: portals into pigmentation genes and ancestry". Trends Genet. 20 (8): 327–32. doi:10.1016/j.tig.2004.06.010. PMID 15262401.
{{cite journal}}: Invalid|ref=harv(help) - ↑ 42.0 42.1 Pearson H (2006). "Genetics: what is a gene?". Nature. 441 (7092): 398–401. Bibcode:2006Natur.441..398P. doi:10.1038/441398a. PMID 16724031.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Visscher PM, Hill WG, Wray NR (2008). "Heritability in the genomics era—concepts and misconceptions". Nat. Rev. Genet. 9 (4): 255–66. doi:10.1038/nrg2322. PMID 18319743.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ 44.0 44.1 Futuyma, Douglas J. (2005). Evolution. Sunderland, Massachusetts: Sinauer Associates, Inc. ISBN 0-87893-187-2.
- ↑ Phillips PC (2008). "Epistasis—the essential role of gene interactions in the structure and evolution of genetic systems". Nat. Rev. Genet. 9 (11): 855–67. doi:10.1038/nrg2452. PMC 2689140. PMID 18852697.
{{cite journal}}: Invalid|ref=harv(help) - ↑ 46.0 46.1 Wu R, Lin M (2006). "Functional mapping – how to map and study the genetic architecture of dynamic complex traits". Nat. Rev. Genet. 7 (3): 229–37. doi:10.1038/nrg1804. PMID 16485021.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Jablonka, E.; Raz, G. (2009). "Transgenerational epigenetic inheritance: Prevalence, mechanisms and implications for the study of heredity and evolution" (PDF). The Quarterly Review of Biology. 84 (2): 131–176. doi:10.1086/598822. PMID 19606595. Archived from the original (PDF) on 2011-07-15. Retrieved 2012-07-21.
- ↑ Harwood AJ; Harwood, J (1998). "Factors affecting levels of genetic diversity in natural populations". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 353 (1366): 177–86. doi:10.1098/rstb.1998.0200. PMC 1692205. PMID 9533122.
{{cite journal}}: Invalid|ref=harv(help) - ↑ 49.0 49.1 Ewens W.J. (2004). Mathematical Population Genetics (2nd Edition). Springer-Verlag, New York. ISBN 0-387-20191-2.
- ↑ Butlin RK, Tregenza T (1998). "Levels of genetic polymorphism: marker loci versus quantitative traits". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 353 (1366): 187–98. doi:10.1098/rstb.1998.0201. PMC 1692210. PMID 9533123.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Wetterbom A, Sevov M, Cavelier L, Bergström TF (2006). "Comparative genomic analysis of human and chimpanzee indicates a key role for indels in primate evolution". J. Mol. Evol. 63 (5): 682–90. doi:10.1007/s00239-006-0045-7. PMID 17075697.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Sawyer SA, Parsch J, Zhang Z, Hartl DL (2007). "Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila". Proc. Natl. Acad. Sci. U.S.A. 104 (16): 6504–10. Bibcode:2007PNAS..104.6504S. doi:10.1073/pnas.0701572104. PMC 1871816. PMID 17409186.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Hastings, P J; Lupski, JR; Rosenberg, SM; Ira, G (2009). "Mechanisms of change in gene copy number". Nature Reviews. Genetics. 10 (8): 551–564. doi:10.1038/nrg2593. PMC 2864001. PMID 19597530.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Carroll SB, Grenier J, Weatherbee SD (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design. Second Edition. Oxford: Blackwell Publishing. ISBN 1-4051-1950-0.
{{cite book}}: More than one of|author=and|last=specified (help)CS1 maint: multiple names: authors list (link) - ↑ Harrison P, Gerstein M (2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". J Mol Biol. 318 (5): 1155–74. doi:10.1016/S0022-2836(02)00109-2. PMID 12083509.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Bowmaker JK (1998). "Evolution of colour vision in vertebrates". Eye (London, England). 12 (Pt 3b): 541–7. doi:10.1038/eye.1998.143. PMID 9775215.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Gregory TR, Hebert PD (1999). "The modulation of DNA content: proximate causes and ultimate consequences". Genome Res. 9 (4): 317–24. doi:10.1101/gr.9.4.317. PMID 10207154.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Hurles M (2004). "Gene duplication: the genomic trade in spare parts". PLoS Biol. 2 (7): E206. doi:10.1371/journal.pbio.0020206. PMC 449868. PMID 15252449.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: unflagged free DOI (link) - ↑ Liu N, Okamura K, Tyler DM (2008). "The evolution and functional diversification of animal microRNA genes". Cell Res. 18 (10): 985–96. doi:10.1038/cr.2008.278. PMC 2712117. PMID 18711447.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Siepel A (2009). "Darwinian alchemy: Human genes from noncoding DNA". Genome Res. 19 (10): 1693–5. doi:10.1101/gr.098376.109. PMC 2765273. PMID 19797681.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Orengo CA, Thornton JM (2005). "Protein families and their evolution-a structural perspective". Annu. Rev. Biochem. 74 (1): 867–900. doi:10.1146/annurev.biochem.74.082803.133029. PMID 15954844.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Long M, Betrán E, Thornton K, Wang W (2003). "The origin of new genes: glimpses from the young and old". Nat. Rev. Genet. 4 (11): 865–75. doi:10.1038/nrg1204. PMID 14634634.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Wang M, Caetano-Anollés G (2009). "The evolutionary mechanics of domain organization in proteomes and the rise of modularity in the protein world". Structure. 17 (1): 66–78. doi:10.1016/j.str.2008.11.008. PMID 19141283.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Radding C (1982). "Homologous pairing and strand exchange in genetic recombination". Annu. Rev. Genet. 16 (1): 405–37. doi:10.1146/annurev.ge.16.120182.002201. PMID 6297377.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Agrawal AF (2006). "Evolution of sex: why do organisms shuffle their genotypes?". Curr. Biol. 16 (17): R696. doi:10.1016/j.cub.2006.07.063. PMID 16950096.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Peters AD, Otto SP (2003). "Liberating genetic variance through sex". BioEssays. 25 (6): 533–7. doi:10.1002/bies.10291. PMID 12766942.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Goddard MR, Godfray HC, Burt A (2005). "Sex increases the efficacy of natural selection in experimental yeast populations". Nature. 434 (7033): 636–40. Bibcode:2005Natur.434..636G. doi:10.1038/nature03405. PMID 15800622.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ 68.0 68.1 Morjan C, Rieseberg L (2004). "How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles". Mol. Ecol. 13 (6): 1341–56. doi:10.1111/j.1365-294X.2004.02164.x. PMC 2600545. PMID 15140081.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Boucher Y, Douady CJ, Papke RT, Walsh DA, Boudreau ME, Nesbo CL, Case RJ, Doolittle WF (2003). "Lateral gene transfer and the origins of prokaryotic groups". Annu Rev Genet. 37 (1): 283–328. doi:10.1146/annurev.genet.37.050503.084247. PMID 14616063.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Walsh T (2006). "Combinatorial genetic evolution of multiresistance". Curr. Opin. Microbiol. 9 (5): 476–82. doi:10.1016/j.mib.2006.08.009. PMID 16942901.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Kondo N, Nikoh N, Ijichi N, Shimada M, Fukatsu T (2002). "Genome fragment of Wolbachia endosymbiont transferred to X chromosome of host insect". Proc. Natl. Acad. Sci. U.S.A. 99 (22): 14280–5. Bibcode:2002PNAS...9914280K. doi:10.1073/pnas.222228199. PMC 137875. PMID 12386340.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Sprague G (1991). "Genetic exchange between kingdoms". Curr. Opin. Genet. Dev. 1 (4): 530–3. doi:10.1016/S0959-437X(05)80203-5. PMID 1822285.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Baldo A, McClure M (September 1, 1999). "Evolution and horizontal transfer of dUTPase-encoding genes in viruses and their hosts". J. Virol. 73 (9): 7710–21. PMC 104298. PMID 10438861.
{{cite journal}}: Invalid|ref=harv(help) - ↑ River, M. C. and Lake, J. A. (2004). "The ring of life provides evidence for a genome fusion origin of eukaryotes". Nature. 431 (9): 152–5. Bibcode:2004Natur.431..152R. doi:10.1038/nature02848. PMID 15356622.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ 75.0 75.1 Hurst LD (2009). "Fundamental concepts in genetics: genetics and the understanding of selection". Nat. Rev. Genet. 10 (2): 83–93. doi:10.1038/nrg2506. PMID 19119264.
{{cite journal}}: Invalid|ref=harv(help) - ↑ 76.0 76.1 76.2 Orr HA (2009). "Fitness and its role in evolutionary genetics". Nat. Rev. Genet. 10 (8): 531–9. doi:10.1038/nrg2603. PMC 2753274. PMID 19546856.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Lande R, Arnold SJ (1983). "The measurement of selection on correlated characters". Evolution. 37 (6): 1210–26. doi:10.2307/2408842. JSTOR 2408842.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Goldberg, Emma E; Igić, B (2008). "On phylogenetic tests of irreversible evolution". Evolution. 62 (11): 2727–2741. doi:10.1111/j.1558-5646.2008.00505.x. PMID 18764918.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Collin, Rachel; Miglietta, MP (2008). "Reversing opinions on Dollo's Law". Trends in Ecology & Evolution. 23 (11): 602–609. doi:10.1016/j.tree.2008.06.013. PMID 18814933.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Hoekstra H, Hoekstra J, Berrigan D, Vignieri S, Hoang A, Hill C, Beerli P, Kingsolver J (2001). "Strength and tempo of directional selection in the wild". Proc. Natl. Acad. Sci. U.S.A. 98 (16): 9157–60. Bibcode:2001PNAS...98.9157H. doi:10.1073/pnas.161281098. PMC 55389. PMID 11470913.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Felsenstein (November 1, 1979). "Excursions along the Interface between Disruptive and Stabilizing Selection". Genetics. 93 (3): 773–95. PMC 1214112. PMID 17248980.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Andersson M, Simmons L (2006). "Sexual selection and mate choice". Trends Ecol. Evol. (Amst.). 21 (6): 296–302. doi:10.1016/j.tree.2006.03.015. PMID 16769428.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Kokko H, Brooks R, McNamara J, Houston A (2002). "The sexual selection continuum". Proc. Biol. Sci. 269 (1498): 1331–40. doi:10.1098/rspb.2002.2020. PMC 1691039. PMID 12079655.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Hunt J, Brooks R, Jennions M, Smith M, Bentsen C, Bussière L (2004). "High-quality male field crickets invest heavily in sexual display but die young". Nature. 432 (7020): 1024–7. Bibcode:2004Natur.432.1024H. doi:10.1038/nature03084. PMID 15616562.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Lynch, M. (2007). "The frailty of adaptive hypotheses for the origins of organismal complexity". PNAS. 104: 8597–8604. Bibcode:2007PNAS..104.8597L. doi:10.1073/pnas.0702207104.
- ↑ Haldane, JBS (1933). "The Part Played by Recurrent Mutation in Evolution". American Naturalist. 67: 5–19. JSTOR 2457127.
- ↑ Protas, Meredith; Conrad, M; Gross, JB; Tabin, C; Borowsky, R (2007). "Regressive evolution in the Mexican cave tetra, Astyanax mexicanus". Current Biology. 17 (5): 452–454. doi:10.1016/j.cub.2007.01.051. PMC 2570642. PMID 17306543.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Masel J (2011). "Genetic drift". Current Biology. 21 (20): R837–R838. doi:10.1016/j.cub.2011.08.007. PMID 22032182.
- ↑ ജനറ്റിക് ഡ്രിഫ്റ്റ്
- ↑ Lien S, Szyda J, Schechinger B, Rappold G, Arnheim N (2000). "Evidence for heterogeneity in recombination in the human pseudoautosomal region: high resolution analysis by sperm typing and radiation-hybrid mapping". Am. J. Hum. Genet. 66 (2): 557–66. doi:10.1086/302754. PMC 1288109. PMID 10677316.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Barton, N H (2000). "Genetic hitchhiking". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 355 (1403): 1553–1562. doi:10.1098/rstb.2000.0716. PMC 1692896. PMID 11127900.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Scott EC, Matzke NJ (2007). "Biological design in science classrooms". Proc. Natl. Acad. Sci. U.S.A. 104 (suppl_1): 8669–76. Bibcode:2007PNAS..104.8669S. doi:10.1073/pnas.0701505104. PMC 1876445. PMID 17494747.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Hendry AP, Kinnison MT (2001). "An introduction to microevolution: rate, pattern, process". Genetica. 112–113: 1–8. doi:10.1023/A:1013368628607. PMID 11838760.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Leroi AM (2000). "The scale independence of evolution". Evol. Dev. 2 (2): 67–77. doi:10.1046/j.1525-142x.2000.00044.x. PMID 11258392.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Michael J. Dougherty. Is the human race evolving or devolving? Scientific American July 20, 1998.
- ↑ TalkOrigins Archive response to Creationist claims – Claim CB932: Evolution of degenerate forms
- ↑ Carroll SB (2001). "Chance and necessity: the evolution of morphological complexity and diversity". Nature. 409 (6823): 1102–9. doi:10.1038/35059227. PMID 11234024.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Whitman W, Coleman D, Wiebe W (1998). "Prokaryotes: the unseen majority". Proc Natl Acad Sci U S A. 95 (12): 6578–83. Bibcode:1998PNAS...95.6578W. doi:10.1073/pnas.95.12.6578. PMC 33863. PMID 9618454.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Nealson K (1999). "Post-Viking microbiology: new approaches, new data, new insights". Orig Life Evol Biosph. 29 (1): 73–93. doi:10.1023/A:1006515817767. PMID 11536899.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Schloss P, Handelsman J (2004). "Status of the microbial census". Microbiol Mol Biol Rev. 68 (4): 686–91. doi:10.1128/MMBR.68.4.686-691.2004. PMC 539005. PMID 15590780.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Mayr, Ernst 1982. The growth of biological thought. Harvard. p483: "Adaptation... could no longer be considered a static condition, a product of a creative past and became instead a continuing dynamic process."
- ↑ The Oxford Dictionary of Science defines adaptation as "Any change in the structure or functioning of an organism that makes it better suited to its environment".
- ↑ Orr H (2005). "The genetic theory of adaptation: a brief history". Nat. Rev. Genet. 6 (2): 119–27. doi:10.1038/nrg1523. PMID 15716908.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Nakajima A, Sugimoto Y, Yoneyama H, Nakae T (2002). "High-level fluoroquinolone resistance in Pseudomonas aeruginosa due to interplay of the MexAB-OprM efflux pump and the DNA gyrase mutation". Microbiol. Immunol. 46 (6): 391–5. PMID 12153116.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Blount ZD, Borland CZ, Lenski RE (2008). "Inaugural Article: Historical contingency and the evolution of a key innovation in an experimental population of Escherichia coli". Proc. Natl. Acad. Sci. U.S.A. 105 (23): 7899–906. Bibcode:2008PNAS..105.7899B. doi:10.1073/pnas.0803151105. PMC 2430337. PMID 18524956.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Okada H, Negoro S, Kimura H, Nakamura S (1983). "Evolutionary adaptation of plasmid-encoded enzymes for degrading nylon oligomers". Nature. 306 (5939): 203–6. Bibcode:1983Natur.306..203O. doi:10.1038/306203a0. PMID 6646204.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Ohno S (1984). "Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence". Proc. Natl. Acad. Sci. U.S.A. 81 (8): 2421–5. Bibcode:1984PNAS...81.2421O. doi:10.1073/pnas.81.8.2421. PMC 345072. PMID 6585807.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Copley SD (2000). "Evolution of a metabolic pathway for degradation of a toxic xenobiotic: the patchwork approach". Trends Biochem. Sci. 25 (6): 261–5. doi:10.1016/S0968-0004(00)01562-0. PMID 10838562.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Crawford RL, Jung CM, Strap JL (2007). "The recent evolution of pentachlorophenol (PCP)-4-monooxygenase (PcpB) and associated pathways for bacterial degradation of PCP". Biodegradation. 18 (5): 525–39. doi:10.1007/s10532-006-9090-6. PMID 17123025.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ 110.0 110.1 Bejder L, Hall BK (2002). "Limbs in whales and limblessness in other vertebrates: mechanisms of evolutionary and developmental transformation and loss". Evol. Dev. 4 (6): 445–58. doi:10.1046/j.1525-142X.2002.02033.x. PMID 12492145.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Young, Nathan M.; Hallgrímsson, B (2005). "Serial homology and the evolution of mammalian limb covariation structure". Evolution. 59 (12): 2691–704. doi:10.1554/05-233.1. PMID 16526515. Retrieved September 24, 2009.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Hall, Brian K (2003). "Descent with modification: the unity underlying homology and homoplasy as seen through an analysis of development and evolution". Biological Reviews of the Cambridge Philosophical Society. 78 (3): 409–433. doi:10.1017/S1464793102006097. PMID 14558591.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Shubin, Neil; Tabin, C; Carroll, S (2009). "Deep homology and the origins of evolutionary novelty". Nature. 457 (7231): 818–823. Bibcode:2009Natur.457..818S. doi:10.1038/nature07891. PMID 19212399.
{{cite journal}}: Invalid|ref=harv(help) - ↑ 114.0 114.1 114.2 Fong D, Kane T, Culver D (1995). "Vestigialization and Loss of Nonfunctional Characters". Ann. Rev. Ecol. Syst. 26 (4): 249–68. doi:10.1146/annurev.es.26.110195.001341.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link) - ↑ Jeffery WR (2005). "Adaptive evolution of eye degeneration in the Mexican blind cavefish". J. Hered. 96 (3): 185–96. doi:10.1093/jhered/esi028. PMID 15653557.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Maxwell EE, Larsson HC (2007). "Osteology and myology of the wing of the Emu (Dromaius novaehollandiae) and its bearing on the evolution of vestigial structures". J. Morphol. 268 (5): 423–41. doi:10.1002/jmor.10527. PMID 17390336.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Zhang Z, Gerstein M (2004). "Large-scale analysis of pseudogenes in the human genome". Curr. Opin. Genet. Dev. 14 (4): 328–35. doi:10.1016/j.gde.2004.06.003. PMID 15261647.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Silvestri AR, Singh I (2003). "The unresolved problem of the third molar: would people be better off without it?". Journal of the American Dental Association (1939). 134 (4): 450–5. PMID 12733778. Archived from the original on 2014-08-23. Retrieved 2013-05-08.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Coyne, Jerry A. (2009). Why Evolution is True. Penguin Group. p. 62. ISBN 978-0-670-02053-9.
- ↑ Darwin, Charles. (1872) The Expression of the Emotions in Man and Animals John Murray, London.
- ↑ Peter Gray (2007). Psychology (fifth ed.). Worth Publishers. p. 66. ISBN 0-7167-0617-2.
- ↑ Coyne, Jerry A. (2009). Why Evolution Is True. Penguin Group. pp. 85–86. ISBN 978-0-670-02053-9.
- ↑ Anthony Stevens (1982). Archetype: A Natural History of the Self. Routledge & Kegan Paul. p. 87. ISBN 0-7100-0980-1.
- ↑ Wade MJ (2007). "The co-evolutionary genetics of ecological communities". Nat. Rev. Genet. 8 (3): 185–95. doi:10.1038/nrg2031. PMID 17279094.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Geffeney S, Brodie ED, Ruben PC, Brodie ED (2002). "Mechanisms of adaptation in a predator-prey arms race: TTX-resistant sodium channels". Science. 297 (5585): 1336–9. Bibcode:2002Sci...297.1336G. doi:10.1126/science.1074310. PMID 12193784.
{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link)
*Brodie ED, Ridenhour BJ, Brodie ED (2002). "The evolutionary response of predators to dangerous prey: hotspots and coldspots in the geographic mosaic of coevolution between garter snakes and newts". Evolution. 56 (10): 2067–82. PMID 12449493.{{cite journal}}: Invalid|ref=harv(help)CS1 maint: multiple names: authors list (link)
*Sean B. Carroll (December 21, 2009). "Remarkable Creatures – Clues to Toxins in Deadly Delicacies of the Animal Kingdom". New York Times. - ↑ Sachs J (2006). "Cooperation within and among species". J. Evol. Biol. 19 (5): 1415–8, discussion 1426–36. doi:10.1111/j.1420-9101.2006.01152.x. PMID 16910971.
{{cite journal}}: Invalid|ref=harv(help)
*Nowak M (2006). "Five rules for the evolution of cooperation". Science. 314 (5805): 1560–3. Bibcode:2006Sci...314.1560N. doi:10.1126/science.1133755. PMC 3279745. PMID 17158317.{{cite journal}}: Invalid|ref=harv(help) - ↑ Paszkowski U (2006). "Mutualism and parasitism: the yin and yang of plant symbioses". Curr. Opin. Plant Biol. 9 (4): 364–70. doi:10.1016/j.pbi.2006.05.008. PMID 16713732.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Hause B, Fester T (2005). "Molecular and cell biology of arbuscular mycorrhizal symbiosis". Planta. 221 (2): 184–96. doi:10.1007/s00425-004-1436-x. PMID 15871030.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Bertram J (2000). "The molecular biology of cancer". Mol. Aspects Med. 21 (6): 167–223. doi:10.1016/S0098-2997(00)00007-8. PMID 11173079.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Reeve HK, Hölldobler B (2007). "The emergence of a superorganism through intergroup competition". Proc Natl Acad Sci U S A. 104 (23): 9736–40. Bibcode:2007PNAS..104.9736R. doi:10.1073/pnas.0703466104. PMC 1887545. PMID 17517608.
{{cite journal}}: Invalid|ref=harv(help) - ↑ Axelrod R, Hamilton W (2005). "The evolution of cooperation". Science. 211 (4489): 1390–6. Bibcode:1981Sci...211.1390A. doi:10.1126/science.7466396. PMID 7466396.
{{cite journal}}: Invalid|ref=harv(help) - ↑ 132.0 132.1 Gavrilets, Sergey (October 2003). "Perspective: models of speciation: what have we learned in 40 years?". Evolution. 57 (10). Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution: 2197–2215. doi:10.1554/02-727. ISSN 0014-3820. PMID 14628909.
- ↑ de Queiroz, Kevin (May 3, 2005). "Ernst Mayr and the modern concept of species". Proc. Natl. Acad. Sci. U.S.A. 102 (Suppl. 1). Washington, D.C.: National Academy of Sciences: 6600–6607. Bibcode:2005PNAS..102.6600D. doi:10.1073/pnas.0502030102. ISSN 0027-8424. PMC 1131873. PMID 15851674.
- ↑ Ereshefsky, Marc (December 1992). "Eliminative pluralism". Philosophy of Science. 59 (4). Chicago, IL: University of Chicago Press: 671–690. doi:10.1086/289701. ISSN 0031-8248. JSTOR 188136.
- ↑ Fraser, Christophe; Alm, Eric J.; Polz, Martin F.; et al. (February 6, 2009). "The Bacterial Species Challenge: Making Sense of Genetic and Ecological Diversity". Science. 323 (5915). Washington, D.C.: American Association for the Advancement of Science: 741–746. Bibcode:2009Sci...323..741F. doi:10.1126/science.1159388. ISSN 0036-8075. PMID 19197054.
- ↑ Short, Roger Valentine (October 1975). "The contribution of the mule to scientific thought". Journal of Reproduction and Fertility. Supplement (23). Society for Reproduction and Fertility: 359–364. ISSN 0449-3087. OCLC 1639439. PMID 1107543.
- ↑ Gross, Briana L.; Rieseberg, Loren H. (May–June 2005). "The Ecological Genetics of Homoploid Hybrid Speciation". Journal of Heredity. 96 (3). Oxford: Oxford University Press on behalf of the American Genetic Association: 241–252. doi:10.1093/jhered/esi026. ISSN 0022-1503. PMC 2517139. PMID 15618301.
- ↑ Vrijenhoek, Robert C. (April 4, 2006). "Polyploid Hybrids: Multiple Origins of a Treefrog Species". Current Biology. 16 (7). Cambridge, MA: Cell Press: R245–R247. doi:10.1016/j.cub.2006.03.005. ISSN 0960-9822. PMID 16581499.
- ↑ Burke, John M.; Arnold, Michael L. (December 2001). "Genetics and the fitness of hybrids". Annual Review of Genetics. 35. Palo Alto, CA: Annual Reviews: 31–52. doi:10.1146/annurev.genet.35.102401.085719. ISSN 0066-4197. PMID 11700276.
- ↑ Rice, William R.; Hostert, Ellen E. (December 1993). "Laboratory Experiments on Speciation: What Have We Learned in 40 Years?". Evolution. 47 (6). Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution: 1637–1653. doi:10.2307/2410209. ISSN 0014-3820.
- Jiggins, Chris D.; Bridle, Jon R. (March 2004). "Speciation in the apple maggot fly: a blend of vintages?". Trends in Ecology & Evolution. 19 (3). Cambridge, MA: Cell Press: 111–114. doi:10.1016/j.tree.2003.12.008. ISSN 0169-5347. PMID 16701238.
- Boxhorn, Joseph (September 1, 1995). "Observed Instances of Speciation". TalkOrigins Archive. Houston, TX: The TalkOrigins Foundation, Inc. Retrieved 2008-12-26.
- Weinberg, James R.; Starczak, Victoria R.; Jörg, Daniele (August 1992). "Evidence for Rapid Speciation Following a Founder Event in the Laboratory". Evolution. 46 (4). Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution: 1214–1220. doi:10.2307/2409766. ISSN 0014-3820. JSTOR 2409766.
- ↑ Herrel, Anthony; Huyghe, Katleen; Vanhooydonck, Bieke; et al. (March 25, 2008). "Rapid large-scale evolutionary divergence in morphology and performance associated with exploitation of a different dietary resource". Proc. Natl. Acad. Sci. U.S.A. 105 (12). Washington, D.C.: National Academy of Sciences: 4792–4795. Bibcode:2008PNAS..105.4792H. doi:10.1073/pnas.0711998105. ISSN 0027-8424. PMC 2290806. PMID 18344323.
- ↑ Losos, Jonathan B.; Warhelt, Kenneth I.; Schoener, Thomas W. (May 1, 1997). "Adaptive differentiation following experimental island colonization in Anolis lizards". Nature. 387 (6628). London: Nature Publishing Group: 70–73. Bibcode:1997Natur.387...70L. doi:10.1038/387070a0. ISSN 0028-0836.
- ↑ Hoskin, Conrad J.; Higgle, Megan; McDonald, Keith R.; Moritz, Craig (October 27, 2005). "Reinforcement drives rapid allopatric speciation". Nature. 437 (7063). London: Nature Publishing Group: 1353–1356. Bibcode:2005Natur.437.1353H. doi:10.1038/nature04004. ISSN 0028-0836. PMID 16251964.
- ↑ Templeton, Alan R. (April 1980). "The Theory of Speciation VIA the Founder Principle". Genetics. 94 (4). Bethesda, MD: Genetics Society of America: 1011–1038. ISSN 0016-6731. PMC 1214177. PMID 6777243. Retrieved 2014-12-29.
- ↑ Antonovics, Janis (July 2006). "Evolution in closely adjacent plant populations X: long-term persistence of prereproductive isolation at a mine boundary". Heredity. 97 (1). London: Nature Publishing Group for The Genetics Society: 33–37. doi:10.1038/sj.hdy.6800835. ISSN 0018-067X. PMID 16639420. Retrieved 2014-12-29.
- ↑ Nosil, Patrik; Crespi, Bernard J.; Gries, Regine; Gries, Gerhard (March 2007). "Natural selection and divergence in mate preference during speciation". Genetica. 129 (3). Dordrecht, the Netherlands: Kluwer Academic Publishers: 309–327. doi:10.1007/s10709-006-0013-6. ISSN 0016-6707. PMID 16900317.
- ↑ Savolainen, Vincent; Anstett, Marie-Charlotte; Lexer, Christian; et al. (May 11, 2006). "Sympatric speciation in palms on an oceanic island". Nature. 441 (7090). London: Nature Publishing Group: 210–213. Bibcode:2006Natur.441..210S. doi:10.1038/nature04566. ISSN 0028-0836. PMID 16467788.
- Barluenga, Marta; Stölting, Kai N.; Salzburger, Walter; et al. (February 9, 2006). "Sympatric speciation in Nicaraguan crater lake cichlid fish". Nature. 439 (7077). London: Nature Publishing Group: 719–23. Bibcode:2006Natur.439..719B. doi:10.1038/nature04325. ISSN 0028-0836. PMID 16467837.
- ↑ Benton, Michael J. (April 7, 1995). "Diversification and extinction in the history of life". Science. 268 (5207): 52–58. Bibcode:1995Sci...268...52B. doi:10.1126/science.7701342. ISSN 0036-8075. PMID 7701342.
- ↑ Raup, David M. (March 28, 1986). "Biological extinction in Earth history". Science. 231 (4745): 1528–1533. Bibcode:1986Sci...231.1528R. doi:10.1126/science.11542058. ISSN 0036-8075. PMID 11542058.
- ↑ 150.0 150.1 Raup, David M. (July 19, 1994). "The role of extinction in evolution". Proc. Natl. Acad. Sci. U.S.A. 91 (15): 6758–6763. Bibcode:1994PNAS...91.6758R. doi:10.1073/pnas.91.15.6758. ISSN 0027-8424. PMC 44280. PMID 8041694.
- ↑ Novacek, Michael J.; Cleland, Elsa E. (May 8, 2001). "The current biodiversity extinction event: scenarios for mitigation and recovery". Proc. Natl. Acad. Sci. U.S.A. 98 (10): 5466–5470. Bibcode:2001PNAS...98.5466N. doi:10.1073/pnas.091093698. ISSN 0027-8424. PMC 33235. PMID 11344295.
- ↑ Pimm, Stuart; Raven, Peter; Peterson, Alan; et al. (July 18, 2006). "Human impacts on the rates of recent, present and future bird extinctions". Proc. Natl. Acad. Sci. U.S.A. 103 (29): 10941–10946. Bibcode:2006PNAS..10310941P. doi:10.1073/pnas.0604181103. ISSN 0027-8424. PMC 1544153. PMID 16829570.
- Barnosky, Anthony D.; Koch, Paul L.; Feranec, Robert S.; et al. (October 1, 2004). "Assessing the Causes of Late Pleistocene Extinctions on the Continents". Science. 306 (5693): 70–75. Bibcode:2004Sci...306...70B. CiteSeerX 10.1.1.574.332. doi:10.1126/science.1101476. ISSN 0036-8075. PMID 15459379.
- ↑ Lewis, Owen T. (January 29, 2006). "Climate change, species–area curves and the extinction crisis". Philosophical Transactions of the Royal Society B: Biological Sciences. 361 (1465): 163–171. doi:10.1098/rstb.2005.1712. ISSN 0962-8436. PMC 1831839. PMID 16553315.
- ↑ 154.0 154.1 ലുവ പിഴവ് ഘടകം:Footnotes-ൽ 80 വരിയിൽ : bad argument #1 to 'ipairs' (table expected, got nil)
- ↑ 155.0 155.1 Novacek, Michael J. (November 8, 2014). "Prehistory's Brilliant Future". The New York Times. New York: The New York Times Company. ISSN 0362-4331. Archived from the original on 2014-12-29. Retrieved 2014-12-25.
- ↑ "Researchers find that Earth may be home to 1 trillion species". National Science Foundation. Arlington County, Virginia. May 2, 2016. Archived from the original on 2016-05-04. Retrieved 2016-05-06.
- ↑ Gould, Stephen Jay (February 28, 1998). "Gulliver's further travels: the necessity and difficulty of a hierarchical theory of selection". Philosophical Transactions of the Royal Society B: Biological Sciences. 353 (1366): 307–314. doi:10.1098/rstb.1998.0211. ISSN 0962-8436. PMC 1692213. PMID 9533127.
- ↑ Kutschera, Ulrich; Niklas, Karl J. (June 2004). "The modern theory of biological evolution: an expanded synthesis". Naturwissenschaften. 91 (6): 255–276. Bibcode:2004NW.....91..255K. doi:10.1007/s00114-004-0515-y. ISSN 1432-1904. PMID 15241603.
- ↑ Jablonski, David (May 8, 2001). "Lessons from the past: Evolutionary impacts of mass extinctions". Proc. Natl. Acad. Sci. U.S.A. 98 (10): 5393–5398. Bibcode:2001PNAS...98.5393J. doi:10.1073/pnas.101092598. ISSN 0027-8424. PMC 33224. PMID 11344284.
- ↑ "Age of the Earth". United States Geological Survey. July 9, 2007. Archived from the original on 2005-12-23. Retrieved 2015-05-31.
- ↑ ലുവ പിഴവ് ഘടകം:Footnotes-ൽ 80 വരിയിൽ : bad argument #1 to 'ipairs' (table expected, got nil)
- ↑ Manhesa, Gérard; Allègre, Claude J.; Dupréa, Bernard; Hamelin, Bruno (May 1980). "Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics". Earth and Planetary Science Letters. 47 (3): 370–382. Bibcode:1980E&PSL..47..370M. doi:10.1016/0012-821X(80)90024-2. ISSN 0012-821X.
- ↑ Schopf, J. William; Kudryavtsev, Anatoliy B.; Czaja, Andrew D.; Tripathi, Abhishek B. (October 5, 2007). "Evidence of Archean life: Stromatolites and microfossils". Precambrian Research. 158 (3–4): 141–155. Bibcode:2007PreR..158..141S. doi:10.1016/j.precamres.2007.04.009. ISSN 0301-9268.
- ↑ ലുവ പിഴവ് ഘടകം:Footnotes-ൽ 80 വരിയിൽ : bad argument #1 to 'ipairs' (table expected, got nil)
- ↑ Borenstein, Seth (November 13, 2013). "Oldest fossil found: Meet your microbial mom". Excite. Yonkers, New York: Mindspark Interactive Network. Associated Press. Archived from the original on June 29, 2015. Retrieved 2015-05-31.
- ↑ Pearlman, Jonathan (November 13, 2013). "'Oldest signs of life on Earth found'". The Daily Telegraph. London: Telegraph Media Group. Archived from the original on 2014-12-16. Retrieved 2014-12-15.
- ↑ Noffke, Nora; Christian, Daniel; Wacey, David; Hazen, Robert M. (November 16, 2013). "Microbially Induced Sedimentary Structures Recording an Ancient Ecosystem in the ca. 3.48 Billion-Year-Old Dresser Formation, Pilbara, Western Australia". Astrobiology. 13 (12): 1103–1124. Bibcode:2013AsBio..13.1103N. doi:10.1089/ast.2013.1030. ISSN 1531-1074. PMC 3870916. PMID 24205812.
- ↑ ലുവ പിഴവ് ഘടകം:Footnotes-ൽ 80 വരിയിൽ : bad argument #1 to 'ipairs' (table expected, got nil)
- ↑ Mora, Camilo; Tittensor, Derek P.; Adl, Sina; et al. (August 23, 2011). "How Many Species Are There on Earth and in the Ocean?". PLOS Biology. 9 (8): e1001127. doi:10.1371/journal.pbio.1001127. ISSN 1545-7885. PMC 3160336. PMID 21886479.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ↑ ലുവ പിഴവ് ഘടകം:Footnotes-ൽ 80 വരിയിൽ : bad argument #1 to 'ipairs' (table expected, got nil)
- ↑ ലുവ പിഴവ് ഘടകം:Footnotes-ൽ 80 വരിയിൽ : bad argument #1 to 'ipairs' (table expected, got nil)
- ↑ Roskov, Y.; Abucay, L.; Orrell, T.; Nicolson, D.; et al., eds. (2016). "Species 2000 & ITIS Catalogue of Life, 2016 Annual Checklist". Species 2000. Leiden, Netherlands: Naturalis Biodiversity Center. ISSN 2405-884X. Archived from the original on 2016-11-12. Retrieved 2016-11-06.
- ↑ Doolittle, W. Ford (February 2000). "Uprooting the Tree of Life" (PDF). Scientific American. 282 (2): 90–95. Bibcode:2000SciAm.282b..90D. doi:10.1038/scientificamerican0200-90. ISSN 0036-8733. PMID 10710791. Archived from the original (PDF) on 2006-09-07. Retrieved 2015-04-05.
- ↑ Peretó, Juli (March 2005). "Controversies on the origin of life" (PDF). International Microbiology. 8 (1): 23–31. ISSN 1139-6709. PMID 15906258. Archived from the original (PDF) on 2015-08-24.
- ↑ Joyce, Gerald F. (July 11, 2002). "The antiquity of RNA-based evolution". Nature. 418 (6894): 214–221. Bibcode:2002Natur.418..214J. doi:10.1038/418214a. ISSN 0028-0836. PMID 12110897.
- ↑ Penny, David; Poole, Anthony (December 1999). "The nature of the last universal common ancestor". Current Opinion in Genetics & Development. 9 (6): 672–677. doi:10.1016/S0959-437X(99)00020-9. ISSN 0959-437X. PMID 10607605.
- ↑ Theobald, Douglas L. (May 13, 2010). "A formal test of the theory of universal common ancestry". Nature. 465 (7295): 219–222. Bibcode:2010Natur.465..219T. doi:10.1038/nature09014. ISSN 0028-0836. PMID 20463738.
- ↑ Bapteste, Eric; Walsh, David A. (June 2005). "Does the 'Ring of Life' ring true?". Trends in Microbiology. 13 (6): 256–261. doi:10.1016/j.tim.2005.03.012. ISSN 0966-842X. PMID 15936656.
- ↑ Darwin 1859, p. 1
- ↑ Doolittle, W. Ford; Bapteste, Eric (February 13, 2007). "Pattern pluralism and the Tree of Life hypothesis". Proc. Natl. Acad. Sci. U.S.A. 104 (7): 2043–2049. Bibcode:2007PNAS..104.2043D. doi:10.1073/pnas.0610699104. ISSN 0027-8424. PMC 1892968. PMID 17261804.
- ↑ Kunin, Victor; Goldovsky, Leon; Darzentas, Nikos; Ouzounis, Christos A. (July 2005). "The net of life: Reconstructing the microbial phylogenetic network". Genome Research. 15 (7): 954–959. doi:10.1101/gr.3666505. ISSN 1088-9051. PMC 1172039. PMID 15965028.