ക്രിസ്പർ
ബാക്ടീരിയ, ആർക്കീയ തുടങ്ങിയ പ്രോകാരിയോട്ടുകളുടെ ജീനോമുകളിൽ ആവർത്തനസ്വഭാവമുള്ള ഡിഎൻഎ ശ്രേണികളാണ് ക്രിസ്പർ. ക്ലസ്റ്റേഡ് റെഗുലേർലി ഇന്റർസ്പേസ്ഡ് ഷോർട്ട് പാലിൻഡ്രൊമിക് റിപ്പീറ്റ്സ് എന്നാണ് ഇതിന്റെ പൂർണ്ണരൂപം. [1] വൈറസുകൾ പ്രോകാരിയോട്ടുകളെ ബാധിക്കുമ്പോൾ വൈറസിന്റെ ചില ജീൻ ശ്രേണികൾ പ്രോകാരിയോട്ടുകളുടെ ജീനോമിലെ ആവർത്തനശ്രേണികൾക്കിടയിൽ സ്പേസർ ശ്രേണികളായി അവശേഷിക്കും. വൈറസുകളുടെ ആക്രമണത്തെ പ്രതിരോധിച്ച പ്രോകാരിയോട്ടുകളിൽ ഇത്തരത്തിൽ ആവർത്തിച്ചുകാണപ്പെടുന്ന ജീൻ ശ്രേണികളും സ്പേസർ ശ്രേണികളും ചേർന്നതാണ് ക്രിസ്പർ. പ്രോകാരിയോട്ടുകളുടെ വൈറസ് പ്രതിരോധ സംവിധാനത്തിൽ ഈ ശ്രേണികൾ പ്രധാന പങ്കു വഹിക്കുന്നു.

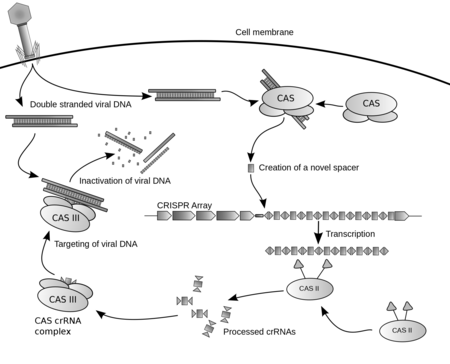
ജീൻ എഡിറ്റിങ് രംഗത്തെ അത്ഭൂതമാണ് ക്രിസ്പർ. ജീൻ എഡിറ്റിങ് നടത്തുമ്പോൾ മുൻകാലങ്ങളിൽ പ്രോകാരിയോട്ടുകളിൽ അവയെ ബാധിച്ച വൈറസുകളുടെ ഡിഎൻഎ അംശം കാണപ്പെടുന്നു. ഈ ഡിഎൻഎ ഉപയോഗിച്ച് സമാന വൈറസുകളിലെ ഡിഎൻഎ ശ്രേണി തിരിച്ചറിയാനും നശിപ്പിക്കാനും ഉപയോഗിക്കുന്നു. അതുകൊണ്ട്ബാക്ടീരിയയുടെ ആക്രമണത്തിന്റെ പ്രതിരോധത്തിനായി ജീൻ എഡിറ്റിങ് മുഖ്യമായ പങ്കുവഹിക്കുന്നുണ്ട്. [2] ഇതിന്റെ അടിസ്ഥാന സാങ്കേതിക വിദ്യയെ ക്രിസ്പർ/കാസ് 9 (CRISPR/Cas9) എന്നറിയപ്പെടുന്നു. ക്രിസ്പർ അനുപാതത്തിന് പരസ്പര പൂരകങ്ങളായ ഡി.എൻ.എ.കളുടെ പ്രത്യേക അംഗങ്ങളെ തിരിച്ചറിയാനും അവ ക്ലിയർ ചെയ്യാനുമുള്ള ഒരു ഗൈഡായി ക്രിസ്പർ സീക്വൻസുകൾ ഉത്പാദിപ്പിക്കുന്ന ഒരു എൻസൈം ആണ് കാസ് 9. ക്രിസ്പർ മെക്കാനിസത്തിൽ പ്രധാന പങ്കുവഹിക്കുന്നത് ഒരു കാസ് പ്രോട്ടീൻ (കാസ് 9) ആണ്. കാസ് 9 നെ കാസ് എൻ 1 (Csn1) എന്നും അറിയപ്പെടുന്നു. ക്രിസ്പർ/കാസ് 9 ജീവികളുടെ ജനിതക വ്യതിയാനത്തിൽ എത്രമാത്രം ഫലപ്രദമാണെന്ന് പ്രത്യേകമായി എടുത്തുപറയേണ്ടതാണ്. [3]
ക്രിസ്പർ/കാസ് സിസ്റ്റത്തിൽ പ്രോകാരിയോട്ടുകളുടെ രോഗപ്രതിരോധവ്യവസ്ഥയിൽ പുറത്തുനിന്നുള്ള അതായത് പ്ലാസ്മിഡ്സിലും, ബാക്ടീരിയോഫേജിലും കാണപ്പെടുന്ന ജനിതക ഘടകങ്ങളെ പ്രതിരോധിക്കുന്നു. [4][5][6] ഇത് അക്വേർഡ് ഇമ്മ്യൂണിറ്റിയ്ക്ക് വഴിതെളിയിക്കുന്നു. ജീൻ എഡിറ്റിങിൽ കാസ് പ്രോട്ടീനുകളെ തിരിച്ചറിയാനും ഡിഎൻഎ മുറിക്കാനും ആർഎൻഎ സഹായിക്കുന്നു. [7] ബാക്ടീരിയയിലെ ജീനിനെ എഡിറ്റിങ് ചെയ്യുമ്പോൾ ഏകദേശം 40% വും ആർക്കീയയിലെ ജീനിനെ എഡിറ്റിങ് ചെയ്യുമ്പോൾ ഏകദേശം 90% വും ക്രിസ്പർ കാണപ്പെടുന്നു. [8]
ക്രിസ്പർ എന്ന പേർ വരുമ്പോൾ ജീൻ എഡിറ്റിങിലെ സ്പേസെർ ജീനുകളുടെ ഉത്ഭവമോ ഉപയോഗമോ ഒന്നും തന്നെ അറിയപ്പെട്ടിരുന്നില്ല. ആ സമയത്ത് ക്രിസ്പറിനെ പ്രോകാരിയോട്ടുകളുടെ ഡിഎൻഎ കളുടെ വിഭാഗങ്ങളായിട്ടാണ് കണ്ടിരുന്നത്. പാലിൻഡ്രോമിക് സീക്വൻസിൽ രണ്ടു ദിശകളിലും ഒരേ ന്യൂക്ലിയോറ്റൈഡുകളായിരിക്കും കാണപ്പെടുന്നത്. ഓരോ ആവർത്തനങ്ങളിലും പിൻതുടരുന്ന സ്പേസെർ ഡിഎൻഎ യുടെ ഓരോ ചെറിയഭാഗങ്ങളും വൈറസ്, പ്ലാസ്മിഡ് എന്നിവയിൽ നിന്നുള്ള ഡിഎൻഎ ആയിരിക്കും. [9] ക്രിസ്പർ സീക്വൻസിൽ ചെറിയ കാസ് സമൂഹം ജീനുകൾ സ്ഥിതിചെയ്യുന്നു.
ക്രിസ്പർ/കാസ് സിസ്റ്റത്തിൽ ക്രിസ്പർ/കാസ് 9 ജീനുകളുടെ എഡിറ്റിങ് വിപുലീകരിക്കുന്നു. കോശങ്ങളിൽ സിന്തറ്റിക് ഗൈഡ് ആർഎൻഎ (gRNA) കാസ് 9 മായി ചേർന്ന് ന്യൂക്ലീസ് എൻസൈം കോപ്ലെക്സ് ഉണ്ടാകുന്നു. കോശങ്ങളിലെ ഈ ജീൻ ഒരു പ്രത്യേക സ്ഥാനത്തുവച്ച് ഡിഎൻഎ മുറിക്കുകയും നിലനിൽക്കുന്ന ബാക്കി ജീനുകളെ കൂട്ടിചേർക്കുകയോ അല്ലെങ്കിൽ പുതിയ ജീനുകളെ കൂട്ടിചേർക്കുകയോ ചെയ്യുന്നു.[10] [11][12]
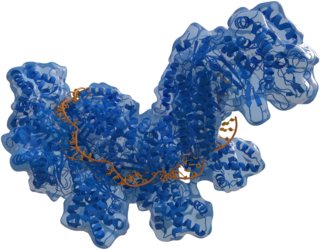
| Cascade (CRISPR-associated complex for antiviral defense) | |
|---|---|
| |
| Structure of crRNA-guided E. coli Cascade complex (Cas, blue) bound to single-stranded DNA (orange). | |
| Identifiers | |
| Organism | |
| Symbol | ? |
| PDB | 4QYZ |
| Other data | |

ചരിത്രം
തിരുത്തുകലോകത്തിന്റെ മൂന്നുഭാഗങ്ങളിലായി ഡിഎൻഎ ക്ലസ്റ്ററുകളുടെ കണ്ടുപിടിത്തം സ്വതന്ത്രമായി ആരംഭിച്ചു. 1987 -ൽ ജപ്പാനിലെ ഒസാക്ക സർവ്വകലാശാലയിലായിരുന്നു ആദ്യത്തെ കണ്ടുപിടിത്തം. ഗവേഷകനായ യോഷിസുമി ഐഷിനോയും അദ്ദേഹത്തിന്റെ സഹപ്രവർത്തകരും ചേർന്ന് അവരുടെ കണ്ടുപിടിത്തങ്ങൾ പ്രസിദ്ധീകരിച്ചു. എഷെറിക്കീയ കോളി ബാക്റ്റീരിയ യുമായി ബന്ധമുള്ള ജീനിലെ സീക്വൻസിനെ ഐപ് ("iap" ) എന്നും വിളിച്ചു.
1993-ൽ നെതർലൻഡിലെ ക്ഷയരോഗ വിദഗ്ദ്ധർ മൈകോബാക്റ്റീറിയം ട്യൂബർകുലോസിസ് ബാക്ടീരിയത്തിൽ ക്ലസ്റ്റർ ഓഫ് ഇന്ററപ്റ്റെഡ് ഡയറക്ട് റിപീറ്റ്സ് (DR) സംബന്ധിച്ച രണ്ട് ലേഖനങ്ങൾ പ്രസിദ്ധീകരിച്ചു. എം. ട്യൂബർകുലോസിസിന് വിവിധ വൈറസുകൾക്കിടയിൽ കാണപ്പെടുന്ന ഡി.ആർ. ഇന്റർവിനിങ് സീക്വൻസിലെ വൈവിധ്യം ഈ ഗവേഷകർ അംഗീകരിച്ചു [13]ടൈപ്പിങ് രീതി രൂപകല്പന ചെയ്യുന്നതിനായി ഈ ഗുണം ഉപയോഗപ്പെടുത്തിയിരുന്നു. സ്പോലിഗോടൈപ്പിംഗ് എന്നിതിനെ വിളിക്കുന്നു. ഇത് ഇന്ന് വരെ ഉപയോഗത്തിലിരിക്കുന്നു.[14][15]
ക്രിസ്പർ മനുഷ്യഭ്രൂണത്തിൽ
തിരുത്തുകക്രിസ്പർ/കാസ് 9 എന്ന സാങ്കേതികവിദ്യ ഉപയോഗിച്ച് മനുഷ്യഭ്രൂണത്തിൽ ആദ്യമായി ജീൻ എഡിറ്റിങ് നടത്തിയത് ചൈനീസ് ശാസ്ത്രജ്ഞനാണ്. പോർട്ട് ലാന്റിലെ ഓറിഗൺ ഹെൽത്ത് ആൻഡ് സർവ്വകലാശാലയിൽ ഷൗഖ്ററ്റ് മിതാലിപോവ് [16]എന്ന പ്രത്യൂൽപ്പാദന ജീവശാസ്ത്രജ്ഞന്റെ നേതൃത്വത്തിലുള്ള ഗവേഷകർ തന്മാത്രാ കത്രികപോലെ പ്രവർത്തിക്കുന്ന ക്രിസ്പർ സാങ്കേതിക വിദ്യയിൽ കാസ് 9 എന്ന പ്രോട്ടീൻ നിശ്ചിതഭാഗത്ത് ഡിഎൻഎ മുറിക്കാൻ സഹായിക്കുന്നു. ഈ പ്രോട്ടീനിനെ നിശ്ചിതഭാഗത്ത് എത്തിക്കുന്ന ഒരു വഴികാട്ടി ആർഎൻഎ ആണ്. [17]
അവലംബം
തിരുത്തുക- ↑ Sawyer E (9 February 2013). "Editing Genomes with the Bacterial Immune System". Scitable. Nature Publishing Group. Retrieved 6 April 2015.
- ↑ Barrangou R (2015). "The roles of CRISPR-Cas systems in adaptive immunity and beyond". Current Opinion in Immunology. 32: 36–41. doi:10.1016/j.coi.2014.12.008. PMID 25574773.
- ↑ Zhang F, Wen Y, Guo X (2014). "CRISPR/Cas9 for genome editing: progress, implications and challenges". Human Molecular Genetics. 23 (R1): R40–6. doi:10.1093/hmg/ddu125. PMID 24651067.
- ↑ Redman M, King A, Watson C, King D (August 2016). "What is CRISPR/Cas9?". Archives of Disease in Childhood. Education and Practice Edition. 101 (4): 213–5. doi:10.1136/archdischild-2016-310459. PMC 4975809 Freely accessible. PMID 27059283.
- ↑ Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, et al. (March 2007). "CRISPR provides acquired resistance against viruses in prokaryotes". Science. 315 (5819): 1709–12. Bibcode:2007Sci...315.1709B. doi:10.1126/science.1138140. PMID 17379808. (registration required)
- ↑ Marraffini LA, Sontheimer EJ (December 2008). "CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA". Science. 322 (5909): 1843–5. Bibcode:2008Sci...322.1843M. doi:10.1126/science.1165771. PMC 2695655 Freely accessible. PMID 19095942.
- ↑ Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J (2016). "Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems". Science. 353 (6299): aad5147. doi:10.1126/science.aad5147. PMID 27493190.
- ↑ Grissa I, Vergnaud G, Pourcel C (May 2007). "The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats". BMC Bioinformatics. 8: 172. doi:10.1186/1471-2105-8-172. PMC 1892036 Freely accessible. PMID 17521438.
- ↑ Marraffini LA, Sontheimer EJ (March 2010). "CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea". Nature Reviews Genetics. 11 (3): 181–90. doi:10.1038/nrg2749. PMC 2928866 Freely accessible. PMID 20125085.
- ↑ Ledford H (2015). "CRISPR, the disruptor". Nature. 522 (7554): 20–4. Bibcode:2015Natur.522...20L. doi:10.1038/522020a. PMID 26040877.
- ↑ Snyder B (21 August 2014). "New technique accelerates genome editing process". research news @ Vanderbilt. Nashville, Tennessee: Vanderbilt University.
- ↑ Hendel A, Bak RO, Clark JT, Kennedy AB, Ryan DE, Roy S, Steinfeld I, Lunstad BD, Kaiser RJ, Wilkens AB, Bacchetta R, Tsalenko A, Dellinger D, Bruhn L, Porteus MH (September 2015). "Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells". Nature Biotechnology. 33 (9): 985–9. doi:10.1038/nbt.3290. PMC 4729442 Freely accessible. PMID 26121415.
- ↑ van Soolingen D, de Haas PE, Hermans PW, Groenen PM, van Embden JD (August 1993). "Comparison of various repetitive DNA elements as genetic markers for strain differentiation and epidemiology of Mycobacterium tuberculosis". Journal of Clinical Microbiology. 31 (8): 1987–95. PMC 265684. PMID 7690367.
- ↑ Groenen PM, Bunschoten AE, van Soolingen D, van Embden JD (December 1993). "Nature of DNA polymorphism in the direct repeat cluster of Mycobacterium tuberculosis; application for strain differentiation by a novel typing method". Molecular Microbiology. 10 (5): 1057–65. doi:10.1111/j.1365-2958.1993.tb00976.x. PMID 7934856.
- ↑ Mojica FJ, Montoliu L (2016). "On the Origin of CRISPR-Cas Technology: From Prokaryotes to Mammals". Trends in Microbiology. 24 (10): 811–20. doi:10.1016/j.tim.2016.06.005. PMID 27401123.
- ↑ Moore, Elizabeth Armstrong (September 17, 2014). "Splice of Life". Willamette Week. p. 12. Retrieved March 4, 2015.
- ↑ https://www.nature.com/news/crispr-fixes-disease-gene-in-viable-human-embryos-1.22382
കൂടുതൽ വായനയ്ക്ക്
തിരുത്തുക- Doudna, Jennifer; Mali, Prashant (23 March 2016). CRISPR-Cas: A Laboratory Manual. New York: Cold Spring Harbor Laboratory Press. ISBN 978-1-62182-131-1.
{{cite book}}: Unknown parameter|name-list-format=ignored (|name-list-style=suggested) (help) - Mohanraju P, Makarova KS, Zetsche B, Zhang F, Koonin EV, van der Oost J (August 2016). "Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems". Science. 353 (6299): aad5147. doi:10.1126/science.aad5147. PMID 27493190.
- Sander JD, Joung JK (April 2014). "CRISPR-Cas systems for editing, regulating and targeting genomes". Nature Biotechnology. 32 (4): 347–55. doi:10.1038/nbt.2842. PMC 4022601. PMID 24584096.
- Slaymaker IM, Gao L, Zetsche B, Scott DA, Yan WX, Zhang F (January 2016). "Rationally engineered Cas9 nucleases with improved specificity". Science. 351 (6268): 84–8. doi:10.1126/science.aad5227. PMC 4714946. PMID 26628643.
- Terns RM, Terns MP (March 2014). "CRISPR-based technologies: prokaryotic defense weapons repurposed". Trends in Genetics. 30 (3): 111–8. doi:10.1016/j.tig.2014.01.003. PMC 3981743. PMID 24555991.
- Westra ER, Buckling A, Fineran PC (May 2014). "CRISPR-Cas systems: beyond adaptive immunity". Nature Reviews Microbiology. 12 (5): 317–26. doi:10.1038/nrmicro3241. PMID 24704746.
- Andersson AF, Banfield JF (May 2008). "Virus population dynamics and acquired virus resistance in natural microbial communities". Science. 320 (5879): 1047–50. Bibcode:2008Sci...320.1047A. doi:10.1126/science.1157358. PMID 18497291.
- Hale C, Kleppe K, Terns RM, Terns MP (December 2008). "Prokaryotic silencing (psi)RNAs in Pyrococcus furiosus". RNA. 14 (12): 2572–9. doi:10.1261/rna.1246808. PMC 2590957. PMID 18971321.
- van der Ploeg JR (June 2009). "Analysis of CRISPR in Streptococcus mutans suggests frequent occurrence of acquired immunity against infection by M102-like bacteriophages". Microbiology. 155 (Pt 6): 1966–76. doi:10.1099/mic.0.027508-0. PMID 19383692.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - van der Oost J, Brouns SJ (November 2009). "RNAi: prokaryotes get in on the act". Cell. 139 (5): 863–5. doi:10.1016/j.cell.2009.11.018. PMID 19945373.
- Karginov FV, Hannon GJ (January 2010). "The CRISPR system: small RNA-guided defense in bacteria and archaea". Molecular Cell. 37 (1): 7–19. doi:10.1016/j.molcel.2009.12.033. PMC 2819186. PMID 20129051.
- Pul U, Wurm R, Arslan Z, Geissen R, Hofmann N, Wagner R (March 2010). "Identification and characterization of E. coli CRISPR-cas promoters and their silencing by H-NS". Molecular Microbiology. 75 (6): 1495–512. doi:10.1111/j.1365-2958.2010.07073.x. PMID 20132443.
- Díez-Villaseñor C, Almendros C, García-Martínez J, Mojica FJ (May 2010). "Diversity of CRISPR loci in Escherichia coli". Microbiology. 156 (Pt 5): 1351–61. doi:10.1099/mic.0.036046-0. PMID 20133361.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - Deveau H, Garneau JE, Moineau S (2010). "CRISPR/Cas system and its role in phage-bacteria interactions". Annual Review of Microbiology. 64: 475–93. doi:10.1146/annurev.micro.112408.134123. PMID 20528693.
- Koonin EV, Makarova KS (December 2009). "CRISPR-Cas: an adaptive immunity system in prokaryotes". F1000 Biology Reports. 1: 95. doi:10.3410/B1-95. PMC 2884157. PMID 20556198.
- "The age of the red pen". The Economist. August 22, 2015. ISSN 0013-0613. Retrieved 2015-08-25.